DNA 염기서열 분석
DNA sequencing| 시리즈의 일부 |
| 유전학 |
|---|
 |
DNA 염기 서열 분석은 DNA의 뉴클레오티드 순서인 핵산 서열을 결정하는 과정입니다. 아데닌, 구아닌, 사이토신, 티민의 네 가지 염기의 순서를 결정하는 모든 방법이나 기술을 포함합니다. 신속한 DNA 염기서열 분석법의 등장으로 생물학적, 의학적 연구와 발견이 크게 가속화되었습니다.[1][2]
DNA 염기서열에 대한 지식은 기초 생물학 연구, DNA 유전체 프로젝트 및 의학 진단, 생명공학, 법의학 생물학, 바이러스학 및 생물학 체계학과 같은 수많은 응용 분야에서 필수 불가결한 것이 되었습니다. 건강한 DNA 염기서열과 돌연변이 DNA 염기서열을 비교하면 다양한 암을 포함한 다양한 질병을 진단할 수 [3]있고 항체 레퍼토리를 특성화할 [4]수 있으며 환자 치료를 안내하는 데 사용할 수 있습니다.[5] DNA 염기서열을 빠르게 분석할 수 있는 방법을 통해 더 빠르고 개별화된 의료를 투여하고 더 많은 유기체를 식별하고 분류할 수 있습니다.[4]
현대 DNA 염기서열 분석 기술로 달성된 빠른 염기서열 분석 속도는 인간 유전체 및 많은 동물, 식물 및 미생물 종의 다른 완전한 DNA 염기서열을 포함하여 수많은 유형 및 생물 종의 완전한 DNA 염기서열, 즉 게놈의 염기서열 분석에 중요한 역할을 했습니다.

최초의 DNA 염기서열은 1970년대 초 학계 연구자들이 2차원 크로마토그래피에 기초한 고된 방법을 사용하여 얻어졌습니다. DNA 시퀀서를 이용한 형광 기반 염기서열 분석법이 개발된 [6]후, DNA 염기서열 분석은 더 쉬워지고 수십 배 빨라졌습니다.[7][8]
적용들
DNA 시퀀싱은 개별 유전자, 더 큰 유전적 영역(즉, 유전자 또는 오페론의 군집), 전체 염색체 또는 유기체의 전체 유전체의 서열을 결정하는 데 사용될 수 있습니다. DNA 염기서열 분석은 RNA나 단백질을 (열린 판독틀을 통해) 간접적으로 염기서열 분석하는 가장 효율적인 방법이기도 합니다. 사실 DNA 염기서열 분석은 생물학과 의학, 포렌식, 인류학 등 여러 과학 분야에서 핵심 기술이 되었습니다.
분자생물학
시퀀싱은 분자 생물학에서 유전체와 그들이 암호화하는 단백질을 연구하는 데 사용됩니다. 시퀀싱을 사용하여 얻은 정보를 통해 연구자는 유전자 및 비코딩 DNA(조절 서열 포함)의 변화, 질병 및 표현형과의 연관성 및 잠재적 약물 표적을 식별할 수 있습니다.
진화생물학
DNA는 한 세대에서 다른 세대로 전달된다는 측면에서 정보를 제공하는 거대 분자이기 때문에, DNA 염기서열 분석은 진화 생물학에서 다른 유기체들이 어떻게 연관되어 있고 어떻게 진화했는지를 연구하는 데 사용됩니다. 2021년 2월, 과학자들은 처음으로 이 경우 매머드인 동물의 유해에서 DNA의 염기서열을 분석한 결과, 지금까지 염기서열 분석된 가장 오래된 DNA인 100만 년 이상이 되었다고 보고했습니다.[9][10]
메타게노믹스
메타게노믹스 분야는 물, 하수, 먼지, 공기에서 여과된 잔해 또는 유기체의 면봉 샘플에 존재하는 유기체의 식별을 포함합니다. 특정 환경에 어떤 유기체가 존재하는지 아는 것은 생태학, 역학, 미생물학 및 기타 분야의 연구에 중요합니다. 염기서열 분석을 통해 연구자들은 예를 들어 마이크로바이옴에 어떤 종류의 미생물이 존재할 수 있는지 결정할 수 있습니다.
바이러스학
대부분의 바이러스는 너무 작아서 광학 현미경으로 볼 수 없기 때문에 염기서열 분석은 바이러스를 식별하고 연구하는 바이러스학의 주요 도구 중 하나입니다.[11] 바이러스 유전체는 DNA나 RNA를 기반으로 합니다. RNA 바이러스는 임상 샘플에서 더 빨리 분해되기 때문에 유전체 염기서열 분석에 더 시간에 민감합니다.[12] 전통적인 생어 염기서열 분석과 차세대 염기서열 분석은 기초 및 임상 연구에서 바이러스 염기서열 분석뿐만 아니라 새로운 바이러스 감염증의 진단, 바이러스 병원체의 분자 역학 및 약물 내성 검사에 사용됩니다. 유전자은행에는 230만 개 이상의 독특한 바이러스 서열이 있습니다.[11] 최근 NGS는 바이러스 유전체를 생성하는 가장 일반적인 접근법으로 전통적인 생어를 능가했습니다.[11]
1990년 조류인플루엔자(AI) 발생 당시, 바이러스 염기서열 분석 결과, 인플루엔자 아형은 메추라기와 가금류 간의 재배열을 통해 유래된 것으로 밝혀졌습니다. 이로 인해 홍콩에서는 살아있는 메추리와 가금류를 함께 시장에서 판매하는 것을 금지하는 법안이 제정되었습니다. 바이러스 시퀀싱은 분자 시계 기술을 사용하여 바이러스 발병이 언제 시작되었는지 추정하는 데 사용할 수도 있습니다.[12]
약
의료 기술자는 유전 질환의 위험이 있는지 확인하기 위해 환자의 유전자(또는 이론적으로 완전한 유전체)를 염기서열 분석할 수 있습니다. 이것은 유전자 검사의 한 형태이지만 일부 유전자 검사에는 DNA 염기서열 분석이 포함되지 않을 수 있습니다.
DNA 염기서열 분석은 희귀질환 진단과 치료에도 점점 더 많이 사용되고 있습니다. 희귀 유전질환을 일으키는 유전자가 밝혀지면서 환자에 대한 분자진단이 더욱 주류를 이루게 됩니다. DNA 염기서열 분석을 통해 임상의는 유전 질환을 식별하고, 질병 관리를 개선하고, 생식 상담을 제공하며, 보다 효과적인 치료법을 제공할 수 있습니다.[13]
또한 DNA 염기서열 분석법은 보다 정밀한 항생제 치료를 가능하게 하여 박테리아 집단에서 항생제 내성이 생길 위험을 줄이는 특정 박테리아를 결정하는 데 유용할 수 있습니다.[14][15][16][17][18][19]
법의학적 수사
DNA 염기서열 분석은 법의학적 신원확인[20] 및 친자확인을 위한 DNA 프로파일링 방법과 함께 사용될 수 있습니다. DNA 검사는 궁극적으로 DNA 인쇄물을 조사 중인 것과 연결하기 위해 지난 수십 년 동안 엄청나게 발전했습니다. 지문, 침, 모낭 등에 있는 DNA 패턴은 각각의 살아있는 유기체를 다른 유기체와 독특하게 구분합니다. DNA를 검사하는 것은 DNA 가닥에서 특정 유전체를 검출하여 독특하고 개별화된 패턴을 생성할 수 있는 기술입니다.
4개의 표준기준
DNA의 표준 구조는 티민(T), 아데닌(A), 시토신(C), 구아닌(G)의 네 가지 염기를 가지고 있습니다. DNA 염기서열결정은 DNA 분자에서 이 염기들의 물리적 순서를 결정하는 것입니다. 그러나 분자 내에는 다른 많은 염기들이 존재할 수 있습니다. 일부 바이러스(특히 박테리오파지)에서 시토신은 히드록시 메틸 또는 히드록시 메틸 글루코스 시토신으로 대체될 수 있습니다.[21] 포유류의 DNA에서 메틸기 또는 포스포설페이트를 갖는 변이체 염기가 발견될 수 있습니다.[22][23] 시퀀싱 기술에 따라 특정 변형, 예를 들어 인간에게 흔한 5mC(5메틸 시토신)이 검출될 수도 있고 검출되지 않을 수도 있습니다.[24]
거의 모든 생물에서 DNA는 4개의 표준 염기만을 사용하여 생체 내에서 합성되며, 복제 후에 일어나는 변형은 5개의 메틸 C와 같은 다른 염기를 생성합니다. 그러나 일부 박테리오파지는 비표준 염기를 직접 통합할 수 있습니다.[25]
DNA는 변형 외에도 UV, 산소 라디칼과 같은 환경 인자에 의해 지속적인 공격을 받고 있습니다. PacBio가 이 https://www.pacb.com/publications/direct-detection-and-sequencing-of-damaged-dna-bases/ 에 발표한 바 있지만, 현재 이러한 손상된 염기의 존재는 대부분의 DNA 염기서열 분석 방법으로 감지되지 않습니다.
역사
DNA 구조와 기능의 발견
디옥시리보핵산(DNA)은 1869년 프리드리히 미셰르에 의해 처음으로 발견되고 분리되었지만, DNA가 아닌 단백질이 유전적 청사진을 생명에 가지고 있다고 생각되었기 때문에 수십 년 동안 연구가 덜 되었습니다. Oswald Avery, Colin MacLeod, Maclyn McCarty가 정제된 DNA가 한 종류의 박테리아를 다른 종류로 바꿀 수 있다는 것을 보여주는 일부 실험의 결과로 1944년 이후 상황이 바뀌었습니다. DNA가 세포의 특성을 변형시킬 수 있다는 것을 보여준 것은 이번이 처음이었습니다.
1953년에 제임스 왓슨과 프란시스 크릭은 로잘린드 프랭클린에 의해 연구되고 있는 결정화된 X선 구조에 기초한 DNA 이중 나선 모델을 제안했습니다. 모델에 따르면, DNA는 서로 감겨 있는 두 가닥의 뉴클레오티드로 구성되어 있으며, 수소 결합에 의해 서로 연결되어 서로 반대 방향으로 달리고 있습니다. 각 가닥은 아데닌(A), 시토신(C), 구아닌(G), 티민(T)의 4개의 상보적인 뉴클레오티드로 구성되어 있으며, 한 가닥의 A는 항상 다른 가닥의 T와 쌍을 이루고, C는 항상 G와 쌍을 이룹니다. 그들은 이러한 구조를 통해 각 가닥이 다른 가닥을 재구성하는 데 사용될 수 있다고 제안했는데, 이는 세대 간 유전 정보 전달의 중심이 되는 아이디어입니다.[26]

단백질의 염기서열 분석의 기초는 1955년까지 췌장에서 분비되는 작은 단백질인 인슐린의 모든 아미노산의 염기서열 분석을 완료한 프레드릭 생거의 연구로 처음 세워졌습니다. 이것은 단백질이 유체에 부유된 물질의 무작위 혼합물이 아니라 특정 분자 패턴을 가진 화학적 실체라는 최초의 결정적인 증거를 제공했습니다. 생어의 인슐린 염기서열 분석 성공은 왓슨과 크릭을 포함한 엑스레이 결정학자들에게 자극을 주었는데, 그들은 지금까지 DNA가 세포 내에서 단백질의 형성을 어떻게 지시하는지를 이해하려고 노력하고 있었습니다. 1954년 10월 프레데릭 생어(Frederick Sanger)의 강의에 참석한 직후, 크릭은 DNA의 뉴클레오티드 배열이 단백질의 아미노산 서열을 결정하고, 이것이 다시 단백질의 기능을 결정하는 데 도움이 된다고 주장하는 이론을 개발하기 시작했습니다. 그는 1958년에 이 이론을 발표했습니다.[27]
RNA 염기서열 분석
RNA 시퀀싱은 뉴클레오티드 시퀀싱의 가장 초기 형태 중 하나였습니다. RNA 염기서열 분석의 주요한 특징은 1972년과[28] 1976년에 겐트 대학(Ghent, 벨기에)의 월터 피어스(Walter Fiers)와 그의 동료들이 확인하고 발표한 최초의 완전한 유전자와 박테리오파지 MS2의 완전한 게놈의 염기서열입니다.[29] 전통적인 RNA 염기서열 분석 방법은 cDNA 분자의 생성을 필요로 하며, 이 분자는 염기서열 분석을 거쳐야 합니다.[30]
초기 DNA 염기서열 분석 방법
DNA 염기서열을 결정하는 첫 번째 방법은 1970년 레이 우(Ray Wu)가 코넬 대학에서 세운 위치별 프라이머 확장 전략과 관련이 있습니다.[31] 현재 시퀀싱 계획에서 두드러지게 나타나는 DNA 중합효소 촉매 및 특정 뉴클레오티드 표지화는 람다 파지 DNA의 응집 말단을 시퀀싱하는 데 사용되었습니다.[32][33][34] 1970년에서 1973년 사이에 Wu와 R Padmanabhan 및 동료들은 이 방법을 사용하여 합성 위치별 프라이머를 사용하여 모든 DNA 서열을 결정할 수 있음을 보여주었습니다.[35][36][8] 프레드릭 생어는 그 후 영국 캠브리지의 MRC 센터에서 보다 신속한 DNA 염기서열 분석 방법을 개발하기 위해 이 프라이머 확장 전략을 채택하여 1977년에 "사슬 종결 억제제를 사용한 DNA 염기서열 분석" 방법을 발표했습니다.[37] 하버드 대학교의 월터 길버트와 앨런 맥삼도 "화학적 분해에 의한 DNA 염기서열 분석"을 포함한 염기서열 분석 방법을 개발했습니다.[38][39] 1973년 길버트와 맥삼은 유랑-점 분석으로 알려진 방법을 사용하여 24개의 염기쌍의 순서를 보고했습니다.[40] 재조합 DNA 기술의 동시 발전으로 염기서열 분석의 발전은 바이러스 이외의 출처에서 DNA 샘플을 분리할 수 있게 되었습니다.
전체 유전체 염기서열 분석

최초로 염기서열이 분석된 완전한 DNA 게놈은 1977년 박테리오파지 φX174의 게놈이었습니다. 의학연구위원회 과학자들은 1984년 엡스타인-바 바이러스의 완전한 DNA 염기서열을 해독하여 172,282개의 뉴클레오티드를 함유하고 있음을 발견했습니다. 염기서열의 완성은 바이러스에 대한 사전 유전자 프로파일 지식 없이 이루어졌기 때문에 DNA 염기서열 분석의 중요한 전환점이 되었습니다.[42][8]
1980년대 초 허버트 폴(Herbert Pohl)과 동료들은 전기영동 과정에서 서열화 반응 혼합물의 DNA 분자를 고정화 매트릭스로 전달하기 위한 비방사성 방법을 개발했습니다.[43][44] EU 유전체 염기서열 분석 프로그램의 틀인 효모 Saccharomyces cerevisiae 염색체 II의 완전한 DNA 염기서열 분석에 집중적으로 사용된 GATC Biotech의 DNA 염기서열 분석기 "Direct-Blotting-Electrophoreis-System GATC 1500"의 상용화에 이어,[45] 르로이 E. 캘리포니아 공과대학의 후드의 연구실은 1986년에 최초의 반자동 DNA 염기서열 분석 기계를 발표했습니다.[46] 1987년 Applied Biosystems의 첫 번째 완전 자동화 시퀀싱 머신인 ABI 370의 마케팅과 새로운 형광 라벨링 기술을 사용한 Dupont의 Genesis 2000의[47] 마케팅이 그 뒤를 이었습니다. 1990년까지 미국 국립보건원(NIH)은 Mycoplasma capricolum, Escherichia coli, Caenorhabditis elegans 및 Saccharomyces cerevisiae에 대한 대규모 염기서열 분석 실험을 시작했습니다. 한편, 인간 게놈의 코딩 부분을 포착하기 위한 시도인 크레이그 벤터(Craig Venter)의 연구실에서 발현된 서열 태그(sequence tag)라고 불리는 인간 cDNA 서열의 시퀀싱이 시작되었습니다.[48] 1995년, 벤터, 해밀턴 스미스와 게놈 연구소(TIGR)의 동료들은 자유 생활 유기체인 헤모필루스 인플루엔자균의 첫 번째 완전한 게놈을 발표했습니다. 원형 염색체에는 1,830,137개의 염기가 포함되어 있으며 사이언스[49] 저널에 게재된 것은 처음으로 전체 유전체 샷건 시퀀싱을 사용하여 초기 매핑 노력을 필요로 하지 않게 된 것입니다.
2001년까지 산탄총 염기서열 분석법은 인간 게놈의 초안 서열을 생성하는 데 사용되었습니다.[50][51]
HTS(High Throughput Sequencing) 방법

DNA 염기서열 분석을 위한 몇 가지 새로운 방법이 1990년대 중후반에 개발되었고 2000년까지 상업용 DNA 염기서열 분석기에서 구현되었습니다. 이것들을 함께 "차세대" 또는 "2세대" 시퀀싱 (NGS) 방법이라고 불렀는데, 이것들을 Sanger 시퀀싱을 포함한 이전의 방법들과 구별하기 위해서입니다. 일반적으로 NGS 기술은 1세대 염기서열 분석과 달리 확장성이 뛰어나 한 번에 전체 유전체를 염기서열 분석할 수 있는 것이 특징입니다. 보통 이것은 게놈을 작은 조각으로 조각내고, 무작위로 조각을 샘플링하고, 아래에 설명된 것과 같은 다양한 기술 중 하나를 사용하여 시퀀싱함으로써 달성됩니다. 자동화된 과정에서 여러 개의 조각이 동시에 시퀀싱되기 때문에 전체 게놈이 가능합니다("massive parallel" sequencing이라는 이름을 부여함).
NGS 기술은 연구자들이 건강에 대한 통찰력을 찾을 수 있도록, 인류학자들이 인간의 기원을 조사할 수 있도록, 그리고 "개인화된 의학" 운동을 촉진하고 있습니다. 그러나 오류의 여지도 더 많이 열어두었습니다. 각각의 알고리즘으로 CSI NGS 포털과 같은 온라인 플랫폼에서 종종 컴파일되는 NGS 데이터의 계산 분석을 수행하는 많은 소프트웨어 도구가 있습니다. 한 소프트웨어 패키지 내의 파라미터도 분석 결과를 변경할 수 있습니다. 또한 DNA 염기서열 분석을 통해 생성되는 대량의 데이터는 염기서열 분석을 위한 새로운 방법과 프로그램의 개발도 요구되어 왔습니다. 이러한 과제를 해결하기 위해 NGS 분야에서 표준을 개발하려는 여러 노력이 시도되었으며, 대부분은 개별 실험실에서 발생하는 소규모 노력이었습니다. 가장 최근에는 FDA의 지원을 받는 대규모 노력이 BioCompute 표준에서 절정에 이르렀습니다.
1990년 10월 26일, 로저 치엔, 페피 로스, 마거릿 패너스톡, 앨런 J 존스턴은 DNA 배열(블롯과 단일 DNA 분자)에 제거 가능한 3' 차단제를 사용한 단계적(base-by-base) 시퀀싱을 설명하는 특허를 출원했습니다.[53] 1996년, 폴 니렌(Paul Nyren)과 그의 제자인 스톡홀름 왕립 공과대학교 모스타파 로나기(Mostafa Ronaghi)는 파이로시퀀싱(pyrosequencing) 방법을 발표했습니다.[54]
1997년 4월 1일 파스칼 메이어와 로랑 파리넬리는 DNA 콜로니 시퀀싱을 설명하는 특허를 세계지식재산권기구에 제출했습니다.[55] Roger Tsien et al. 의 "base-by-base" 시퀀싱 방법과 결합된 본 특허에서 설명된 DNA 샘플 준비 및 무작위 표면-중합효소 연쇄반응(PCR) 배열 방법은 Illumina의 Hi-Seq 게놈 시퀀서에서 구현됩니다.
1998년, 워싱턴 대학교의 Phil Green과 Brent Ewing은 광범위하게 채택된 획기적인 분석 기법인 [56]시퀀서 데이터 분석을 위한 그들의 사전 품질 점수에 대해 설명했으며, 이는 여전히 시퀀서 플랫폼의 정확성을 평가하는 가장 일반적인 척도입니다.[57]
Lynx Therapeutics는 2000년에 MPS(Massive Parallel Signature Sequencing)를 발표하고 판매했습니다. 이 방법은 병렬화된 어댑터/결찰 매개 비드 기반 시퀀싱 기술을 통합했으며 최초의 상업적으로 이용 가능한 "차세대" 시퀀싱 방법으로 사용되었지만 DNA 시퀀서는 독립적인 실험실에 판매되지 않았습니다.[58]
기본방법
맥삼-길버트 염기서열 분석
앨런 맥삼(Allan Maxam)과 월터 길버트(Walter Gilbert)는 1977년 DNA의 화학적 변형과 특정 염기에서의 절단에 기초한 DNA 시퀀싱 방법을 발표했습니다.[38] 화학적 염기서열결정이라고도 알려진 이 방법은 더 이상의 복제 없이 이중가닥 DNA의 정제된 샘플을 사용할 수 있게 했습니다. 이 방법의 방사성 표지 사용과 기술적 복잡성은 생어 방법의 개선이 이루어진 후 광범위한 사용을 억제했습니다.
Maxam-Gilbert 염기서열 분석은 DNA의 한쪽 5' 말단에 방사성 표지와 염기서열 분석을 위한 DNA 단편의 정제가 필요합니다. 화학적 처리는 4개의 반응(G, A+G, C, C+T) 각각에서 4개의 뉴클레오티드 염기 중 1개 또는 2개의 작은 비율로 분열을 발생시킵니다. 수정 화학 물질의 농도는 DNA 분자당 평균 하나의 수정을 도입하도록 제어됩니다. 따라서 방사성 표지된 말단에서 각 분자의 첫 번째 "컷" 부위에 이르기까지 일련의 표지된 단편이 생성됩니다. 크기 분리를 위해 아크릴아미드 겔을 변성시키는 데 네 가지 반응의 단편을 나란히 전기영동합니다. 조각을 시각화하기 위해 젤을 X선 필름에 노출시켜 방사선 촬영을 위해 각각 방사성 표지된 DNA 조각에 해당하는 일련의 어두운 밴드를 생성하고 이로부터 서열을 추론할 수 있습니다.[38]
이 방법은 2023년 현재 대부분 오래되었습니다.[59]
연쇄종결방법
Frederick Sanger와 동료들이 1977년에 개발한 체인 터미네이션 방법은 비교적 쉽고 신뢰할 수 있기 때문에 곧 선택 방법이 되었습니다.[37][60] 발명되었을 때, 체인 터미네이터 방법은 맥삼과 길버트 방법보다 독성 화학물질을 적게 사용하고 방사능 양을 적게 사용했습니다. Sanger 방법은 비교하기 쉽기 때문에 곧 자동화되었으며 1세대 DNA 시퀀서에 사용된 방법이었습니다.
생어 시퀀싱은 1980년대부터 2000년대 중반까지 유행했던 방법입니다. 그 기간 동안 형광 표지화, 모세관 전기영동 및 일반 자동화와 같은 기술에서 큰 발전이 있었습니다. 이러한 개발은 훨씬 더 효율적인 시퀀싱을 가능하게 하여 비용을 낮춥니다. 대량생산 형태의 생어법은 2001년 인류 최초의 유전체를 생산한 기술로 유전체학의 시대를 열었습니다. 그러나 10년 후에 근본적으로 다른 접근 방식이 시장에 출시되어 유전체당 비용이 2001년 1억 달러에서 2011년 1만 달러로 낮아졌습니다.[61]
합성에 의한 시퀀싱
합성에 의한 순차적 시퀀싱(sequential sequencing by synthesis, SBS)의 목적은 DNA 중합효소에 의한 뉴클레오티드의 혼입을 검출함으로써 DNA 샘플의 시퀀싱을 결정하는 것입니다. 조작된 중합효소는 DNA의 단일 가닥의 사본을 합성하는 데 사용되며 각 뉴클레오티드의 통합을 모니터링합니다. 합성에 의한 실시간 시퀀싱의 원리는 1993년에[62] 처음 기술되었으며 몇 년 후에 개선 사항이 발표되었습니다.[63] 핵심 부분은 SBS의 모든 구현예에 대해 매우 유사하며 (1) DNA의 증폭(후속 신호의 향상을 위해) 및 서열화될 DNA를 고체 지지체에 부착하는 것, (2) 고체 지지체 상에 단일 가닥 DNA의 생성, (3) 조작된 중합효소를 이용한 뉴클레오티드 혼입 및 (4) 뉴클레오티드 혼입의 실시간 검출 단계 3-4를 반복하고 상기 단계 4에서 얻은 신호로부터 서열을 조립하는 단계. 합성에 의한 실시간 시퀀싱의 이 원리는 454, PacBio, IonTorrent, Illumina 및 MGI를 포함한 거의 모든 대규모 병렬 시퀀싱 기기에 사용되었습니다.
대규모 시퀀싱 및 데노보 시퀀싱
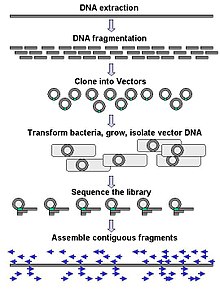
대규모 시퀀싱은 종종 전체 염색체와 같이 매우 긴 DNA 조각의 시퀀싱을 목표로 하지만, 대규모 시퀀싱은 또한 파지 디스플레이에서 발견되는 것과 같은 매우 많은 수의 짧은 시퀀스를 생성하는 데 사용될 수 있습니다. 염색체와 같은 더 긴 표적의 경우, 일반적인 접근법은 큰 DNA 조각을 (제한 효소를 사용하여) 절단하거나 (기계적 힘을 사용하여) 더 짧은 DNA 조각으로 전단하는 것으로 구성됩니다. 그런 다음 단편화된 DNA를 DNA 벡터에 복제하여 대장균과 같은 박테리아 숙주에서 증폭할 수 있습니다. 개별 박테리아 집락에서 정제된 짧은 DNA 단편은 개별적으로 시퀀싱되고 전자적으로 하나의 길고 연속적인 서열로 조립됩니다. 균일한 크기의 DNA 조각을 수집하기 위해 크기 선택 단계를 추가하면 유전체 결합체의 염기서열 분석 효율과 정확도를 향상시킬 수 있다는 연구 결과가 나왔습니다. 이 연구에서 자동 사이징은 수동 젤 사이징보다 더 재현성이 높고 정확한 것으로 입증되었습니다.[64][65][66]
"de novo sequencing"이라는 용어는 특히 이전에 알려진 서열이 없는 DNA의 서열을 결정하는 데 사용되는 방법을 말합니다. 드 노보는 라틴어에서 "처음부터"로 번역합니다. 조립된 시퀀스의 간격은 프라이머 워킹으로 채워질 수 있습니다. 서로 다른 전략은 속도와 정확성에서 서로 다른 절충점을 가지고 있습니다; 샷건 방법은 종종 큰 유전체의 염기서열을 분석하는 데 사용되지만, 특히 염기서열 반복으로 인해 유전체 조립에 종종 격차가 발생하기 때문에 그 조립이 복잡하고 어렵습니다.
대부분의 시퀀싱 접근법은 분자 검출 방법이 단일 분자 시퀀싱에 충분히 민감하지 않기 때문에 개별 DNA 분자를 증폭하기 위해 시험관 내 복제 단계를 사용합니다. 에멀젼 PCR은[67] 프라이머로 코팅된 비드와 함께 개별 DNA 분자를 유성 상 내의 수성 액적에서 분리합니다. 그런 다음 중합효소 연쇄반응(PCR)은 각 비드에 DNA 분자의 클론 사본을 코팅한 다음 나중에 시퀀싱을 위해 고정화합니다. 에멀젼 PCR은 Marguilis et al. (454 Life Sciences에 의해 상용화됨), Sendure and Porreca et al. ("polony sequencing"이라고도 함) 및 SOLiD sequencing (Agentourt, 후에 Applied Biosystems, 현재의 Life Technologies에 의해 개발된)에 의해 개발된 방법에 사용됩니다.[68][69][70] 에멀젼 PCR은 10x Genomics에서 개발한 GemCode 및 Chrome 플랫폼에서도 사용됩니다.[71]
산탄총 염기서열 분석
샷건 시퀀싱은 1000개 이상의 염기쌍, 전체 염색체를 포함하는 DNA 염기서열 분석을 위해 설계된 시퀀싱 방법입니다. 이 방법은 표적 DNA를 무작위 조각으로 분해해야 합니다. 체인 터미네이션 방법을 사용하여 개별 단편을 시퀀싱한 후 중첩된 영역을 기준으로 서열을 재조립할 수 있습니다.[72]
처리율이 높은 방법

차세대 "짧은 읽기" 및 3세대 "긴 읽기" 시퀀싱 방법을 포함하는 고처리량 시퀀싱은 [nt 1]엑솜 시퀀싱, 유전체 시퀀싱, 유전체 재서열화, 전사체 프로파일링(RNA-Seq), DNA-단백질 상호작용(ChIP-sequencing) 및 후성유전체 특성화에 적용됩니다.[73]
저비용 시퀀싱에 대한 높은 요구는 시퀀싱 프로세스를 병렬화하여 수천 개 또는 수백만 개의 시퀀스를 동시에 생성하는 고처리량 시퀀싱 기술의 개발을 주도했습니다.[74][75][76] 고처리량 시퀀싱 기술은 표준 염료 터미네이터 방법으로 가능한 것 이상으로 DNA 시퀀싱 비용을 낮추기 위한 것입니다.[77] 초고처리량 시퀀싱에서는 500,000개에 달하는 합성별 시퀀싱 작업이 병렬로 실행될 수 있습니다.[78][79][80] 그러한 기술들은 단 하루 만에 인간 게놈 전체의 염기서열을 분석할 수 있게 했습니다.[81] 2019년[update] 현재, 고처리량 시퀀싱 제품 개발의 기업 리더로는 일루미나, Qiagen 및 ThermoFisher Scientific이 있습니다.[81]
| 방법 | 읽기길이 | 정확도(단일 판독값이 일치하지 않음) | 실행당 읽기 수 | 실행당 시간 | 10억 베이스당 비용(US$ 단위) | 이점 | 단점들 |
|---|---|---|---|---|---|---|---|
| 단일 분자 실시간 시퀀싱(Pacific Biosciences) | 30,000 bp (N50); | 87%의 원시 읽기 정확도[87] | Sequences 2 SMRT 셀 당 4,000,000개, 100-200 기가바이트[84][88][89] | 30분에서 20시간[84][90] | $7.2-$43.3 | Fast. Detects 4mC, 5mC, 6mA.[91] | 적당한 처리량. 장비는 매우 비쌀 수 있습니다. |
| 이온 반도체(Ion Torrent sequencing) | 600bp까지[92] | 99.6%[93] | 8천만까지 | 2시간 | $66.8-$950 | 저렴한 장비. 빠른. | 호모폴리머 오류. |
| 파이로시퀀싱(454) | 700bp | 99.9% | 백만 | 24시간이 모자라 | $10,000 | 긴 읽기 크기입니다. 빠른. | 런은 비쌉니다. 호모폴리머 오류. |
| 합성에 의한 시퀀싱(Illumina) | MiniSeq, NextSeq: 75-300bp; MiSeq: 50–600 bp; HiSeq 2500: 50-500bp; HiSeq 3/4000: 50–300 bp; HiSeq X: 300 bp | 99.9% (Phred30) | MiniSeq/MiSeq: 1-25백만; NextSeq: 1억 3천만명; HiSeq 2500: 3억 - 20억; HiSeq 3/4000 2.5 billion; HiSeq X: 30억 | 시퀀서 및 지정된 읽기 길이에[94] 따라 1~11일 | $5 ~ $150 | 시퀀서 모델과 원하는 용도에 따라 높은 시퀀스 수율 가능성이 있습니다. | 장비는 매우 비쌀 수 있습니다. 고농도의 DNA가 필요합니다. |
| 복합 프로브 앵커 합성(cPAS-BGI/MGI) | BGISEQ-50: 35-50bp; MGISEQ 200: 50-200bp; BGISEQ-500, MGISEQ-2000: 50-300bp[95] | 99.9% (Phred30) | BGISEQ-50: 160M; MGISEQ 200: 300M; BGISEQ-500: 플로우 셀 당 1300M; MGISEQ-2000: 플로우 셀 당 375M FCS 플로우 셀, 1500M FCL 플로우 셀. | 기기, 읽기 길이 및 한번에 실행되는 플로우 셀의 수에 따라 1~9일이 소요됩니다. | $5– $120 | ||
| 결찰별 시퀀싱(SOLiD sequencing) | 50+35 또는 50+50bp | 99.9% | 12억에서 14억 | 1~2주 | $60–130 | 기본당 비용이 저렴합니다. | 다른 방법보다 느립니다. 회문 배열 순서에 문제가 있습니다.[96] |
| 나노포어 시퀀싱 | 장치가 아닌 라이브러리 준비에 따라 사용자가 읽기 길이(최대 2,272,580bp 보고)를[97] 선택합니다. | ~92~97%의 단일 읽기 | 사용자가 선택한 읽기 길이에 따라 달라집니다. | 실시간으로 스트리밍되는 데이터. 1분에서 48시간 선택 | $7–100 | 가장 긴 개인 읽기입니다. 접근 가능한 사용자 커뮤니티입니다. 휴대용(팜 사이즈)입니다. | 다른 기계보다 낮은 처리량, 90년대 단일 읽기 정확도입니다. |
| GenapSys 시퀀싱 | 약 150bp 싱글엔드 | 99.9% (Phred30) | 100만~1600만 | 24시간 정도 | $667 | 저렴한 기기 비용($10,000) | |
| 체인 터미네이션(Sanger sequencing) | 400~900bp | 99.9% | 해당 없음 | 20분~3시간 | $2,400,000 | 많은 응용 프로그램에 유용합니다. | 대규모 시퀀싱 프로젝트의 경우 비용이 더 많이 들고 비현실적입니다. 이 방법은 또한 플라스미드 복제 또는 PCR의 시간 소모적인 단계를 필요로 합니다. |
긴 읽기 순서법
단일 분자 실시간(SMRT) 시퀀싱
SMRT 시퀀싱은 합성 접근법에 의한 시퀀싱을 기반으로 합니다. DNA는 우물 바닥에 포획 도구가 있는 작은 우물 모양의 용기인 ZMW(제로 모드 도파관)에서 합성됩니다. 시퀀싱은 변형되지 않은 중합효소(ZMW 바닥에 부착)와 용액에서 자유롭게 흐르는 형광 표지된 뉴클레오티드를 사용하여 수행됩니다. 웰은 웰 바닥에서 발생하는 형광만 감지되는 방식으로 구성됩니다. 형광 표지는 DNA 가닥에 결합되면 뉴클레오티드에서 분리되어 변형되지 않은 DNA 가닥을 남깁니다. SMRT 기술 개발업체인 Pacific Biosciences(PacBio)에 따르면, 이 방법은 뉴클레오티드 변형(시토신 메틸화와 같은)을 감지할 수 있게 해줍니다. 이것은 중합효소 동역학의 관찰을 통해 발생합니다. 이 접근 방식은 평균 읽기 길이가 5킬로베이스인 20,000개 이상의 뉴클레오티드를 읽을 수 있습니다.[88][98] 2015년 Pacific Biosciences는 PacBio RS II 기기의 15만 ZMW에 비해 100만 ZMW를 가진 Sequel System이라는 새로운 시퀀싱 기기의 출시를 발표했습니다.[99][100] SMRT 시퀀싱은 "3세대" 또는 "롱 리드" 시퀀싱이라고 합니다.
나노포어 DNA 염기서열 분석
나노포어를 통과하는 DNA는 이온 전류를 변화시킵니다. 이 변화는 DNA 서열의 모양, 크기 및 길이에 따라 달라집니다. 뉴클레오티드의 각 유형은 다른 시간 동안 기공을 통한 이온 흐름을 차단합니다. 이 방법은 변형된 뉴클레오티드가 필요하지 않고 실시간으로 수행됩니다. 나노포어 시퀀싱은 SMRT 시퀀싱과 함께 "3세대" 또는 "롱 리드" 시퀀싱이라고 합니다.
이 방법에 대한 초기 산업 연구는 '엑소뉴클레아제 시퀀싱'이라는 기술에 기반을 두었는데, 여기서 뉴클레오티드가 사이클로덱스트린과 공유 결합된 알파(α)-용혈소 기공에 의해 통과되면서 전기 신호의 판독이 발생했습니다.[101] 하지만 이후 상용화된 방법인 '가닥 염기서열 분석'은 온전한 가닥에서 DNA 염기서열을 분석했습니다.
개발 중인 나노포어 시퀀싱의 두 가지 주요 분야는 고체 상태 나노포어 시퀀싱과 단백질 기반 나노포어 시퀀싱입니다. 단백질 나노포어 시퀀싱은 α-용혈소, MspA(Mycobacterium smegmatis Porin A) 또는 CssG와 같은 막 단백질 복합체를 활용하며, 이는 뉴클레오티드의 개별 및 그룹을 구별하는 능력을 감안할 때 큰 가능성을 보여줍니다.[102] 이에 비해 고체 나노포어 시퀀싱은 질화규소, 산화알루미늄 등 합성소재를 활용하며 기계적 능력과 열적, 화학적 안정성이 우수해 선호됩니다.[103] 나노포어 어레이는 직경이 8 나노미터보다 작은 수백 개의 기공을 포함할 수 있기 때문에 이러한 유형의 시퀀싱에는 제조 방법이 필수적입니다.[102]
단일 가닥의 DNA나 RNA 분자는 8나노미터 이하의 생물학적 기공을 통해 엄격한 선형 배열로 전기영동적으로 구동될 수 있고, 분자가 기공을 통해 이동하면서 이온 전류를 방출하는 것을 고려할 때 이를 감지할 수 있다는 아이디어에서 비롯되었습니다. 포어는 서로 다른 염기를 인식할 수 있는 검출 영역을 포함하고 있으며, 각 염기는 포어를 통과할 때 염기 서열에 해당하는 다양한 시간 특이적 신호를 생성하여 평가됩니다.[103] 성공을 위해서는 기공을 통한 DNA 수송을 정확하게 제어하는 것이 중요합니다. 엑소뉴클레아제, 중합효소 등 다양한 효소를 이용하여 기공의 입구 근처에 위치시켜 이 과정을 조절해 왔습니다.[104]
단독자 시퀀싱 방법
MPS(Massive Parallel Signature Sequencing)
최초의 처리율이 높은 시퀀싱 기술인 MPSS(Massive Parallel Signature Sequencing)는 Sydney Brenner와 Sam Eletr이 1992년에 설립한 회사인 Lynx Therapeutics에서 1990년대에 개발되었습니다. MPSS는 어댑터 결찰에 이어 어댑터 디코딩의 복잡한 접근 방식을 사용하여 4개의 뉴클레오티드를 증분하여 시퀀스를 판독하는 비드 기반 방법이었습니다. 이 방법은 특정 서열의 편향 또는 손실에 취약하도록 만들었습니다. 기술이 워낙 복잡했기 때문에 MPSS는 링스 테라퓨틱스가 '사내'로 수행했을 뿐 DNA 염기서열 분석기는 독립된 실험실에 판매되지 않았습니다. Lynx Therapeutics는 2004년 Solexa(이후 Illumina에 인수됨)와 합병하여 MPSS를 쓸모없게 만든 Manteia Predictive Medicine에서 획득한 더 간단한 접근법인 합성별 시퀀싱의 개발로 이어졌습니다. 그러나 MPSS 출력의 본질적인 특성은 수십만 개의 짧은 DNA 서열을 포함하여 나중의 고처리량 데이터 유형의 전형이었습니다. MPSS의 경우 유전자 발현 수준 측정을 위한 cDNA 시퀀싱에 일반적으로 사용되었습니다.[58]
폴리니 시퀀싱
조지 M의 실험실에서 개발된 폴리니 시퀀싱 방법. Church at Harvard는 최초의 고처리량 시퀀싱 시스템 중 하나였으며 2005년에 전체 대장균 유전체의 염기서열을 분석하는 데 사용되었습니다.[105] 시험관 내 페어드 태그 라이브러리와 에멀젼 PCR, 자동 현미경 및 결찰 기반 시퀀싱 화학을 결합하여 >99.9999%의 정확도와 생어 시퀀싱의 약 1/9 비용으로 대장균 유전체를 시퀀싱했습니다.[105] 이 기술은 Agentcourt Biosciences에 라이선스되었고, 그 후 Agentcourt Personal Genomics로 분사되었으며, 결국 Applied Biosystems SOLiD 플랫폼에 통합되었습니다. Applied Biosystems는 나중에 현재 Thermo Fisher Scientific의 일부인 Life Technologies에 인수되었습니다.
454 파이로시퀀싱
파이로시퀀싱의 병렬화된 버전은 454 생명과학에 의해 개발되었고, 그 후 로슈 진단에 의해 인수되었습니다. 이 방법은 오일 용액(에멀젼 PCR)에서 물방울 내부의 DNA를 증폭하고, 각 물방울은 단일 프라이머 코팅된 비드에 단일 DNA 템플릿을 포함하고 그 다음 클론 집락을 형성합니다. 시퀀싱 머신은 단일 비드 및 시퀀싱 효소를 각각 포함하는 많은 피콜리터-부피 웰을 포함합니다. 파이로시퀀싱은 루시퍼라제를 사용하여 초기 DNA에 추가된 개별 뉴클레오티드의 검출을 위한 빛을 생성하고 결합된 데이터는 서열 판독을 생성하는 데 사용됩니다.[68] 이 기술은 한쪽 끝은 생어 시퀀싱, 다른 쪽 끝은 솔렉사와 솔리드에 비해 기본당 중간 읽기 길이와 가격을 제공합니다.[77]
일루미나(솔렉사) 시퀀싱
현재 일루미나의 일부인 솔렉사는 1998년 샹카르 발라수브라마니안과 데이비드 클레너먼이 설립해 가역적인 염료 종결자 기술과 엔지니어링 폴리머라제를 기반으로 한 시퀀싱 방법을 개발했습니다.[106] 가역적인 종결 화학 개념은 파리에 있는 파스퇴르 연구소의 브루노 카나드와 시몬 사르파티에 의해 발명되었습니다.[107][108] 관련 특허에 이름을 올린 사람들이 솔렉사에서 내부적으로 개발했습니다. 2004년, Solexa는 Pascal Mayer와 Laurent Farinelli가 1997년에 발명한 대규모 병렬 시퀀싱 기술을 얻기 위해 Manteia Predictive Medicine이라는 회사를 인수했습니다.[55] DNA 군집 또는 DNA 군집에 기반을 두고 있으며, 이는 표면에서 DNA의 클론 증폭을 포함합니다. 클러스터 기술은 캘리포니아의 Lynx Therapeutics와 공동 인수했습니다. Solexa Ltd.는 나중에 Lynx와 합병하여 Solexa Inc.를 설립했습니다.


이 방법은 먼저 DNA 분자와 프라이머를 슬라이드나 유동 세포에 부착하고 중합효소로 증폭시켜 나중에 만들어진 'DNA 클러스터'인 국소 클론 DNA 집락이 형성되도록 합니다. 서열을 결정하기 위해 4가지 유형의 가역적 터미네이터 염기(RT-base)가 추가되고 비통합 뉴클레오티드가 세척됩니다. 카메라는 형광 표지된 뉴클레오티드의 이미지를 촬영합니다. 그런 다음, 말단 3' 차단제와 함께 염료를 DNA에서 화학적으로 제거하여 다음 주기를 시작할 수 있습니다. 파이로시퀀싱과 달리 DNA 사슬은 한 번에 하나의 뉴클레오티드로 확장되고 이미지 획득이 지연되는 순간에 수행될 수 있으므로 매우 큰 배열의 DNA 집락을 단일 카메라에서 찍은 순차적 이미지로 캡처할 수 있습니다.

효소 반응과 이미지 캡처를 분리하면 최적의 처리량과 이론적으로 제한 없는 시퀀싱 용량이 가능합니다. 따라서 최적의 구성으로 궁극적으로 도달할 수 있는 기기 처리량은 카메라의 아날로그-디지털 변환 속도에만 따라 결정되며, 카메라 수에 카메라 수를 곱하고 최적으로 시각화하는 데 필요한 DNA 집락당 픽셀 수로 나뉩니다(약 10 픽셀/집락). 2012년에는 카메라가 10 MHz 이상의 A/D 변환 속도로 작동하고 광학, 유체학 및 효소학을 이용할 수 있으므로 처리량은 초당 100만 개의 뉴클레오티드의 배수가 될 수 있으며, 이는 기기당 시간당 1배의 커버리지로 약 1개의 인간 유전체에 해당하며, 1개의 인간 유전체가 재시퀀싱됩니다(약 1개). 기기당 하루 30배)입니다(단일 카메라 장착).[109]
복합 프로브 앵커 합성(cPAS)
이 방법은 2013년 중국 유전체학 회사 BGI의 일부가 된 Complete Genomics가[110] 설명한 cPAL(조합 프로브 앵커 결찰 기술)을 업그레이드한 것입니다.[111] 두 회사는 더 긴 읽기 길이, 반응 시간 단축 및 결과 도출 시간 단축을 위해 기술을 개선했습니다. 또한 데이터는 이제 표준 FASTQ 파일 형식에서 연속적인 전체 길이 읽기로 생성되며 대부분의 짧은 읽기 기반 생물정보학 분석 파이프라인에서 그대로 사용할 수 있습니다.[112][citation needed]
이 고처리량 시퀀싱 기술의 기반을 이루는 두 가지 기술은 DNA 나노볼(DNB)과 나노볼을 고체 표면에 부착하기 위한 패턴 어레이입니다.[110] DNA 나노볼은 이중 가닥, 어댑터 연결 라이브러리를 변성시키고 전방 가닥을 스플린트 올리고뉴클레오티드에만 연결하여 ssDNA 원을 형성함으로써 간단히 형성됩니다. DNA 삽입물을 포함하는 원의 충실한 사본은 약 300-500부를 생성하는 Rolling Circle Amplification을 사용하여 생성됩니다. ssDNA의 긴 가닥이 스스로 접혀 지름이 약 220nm인 3차원 나노볼 구조를 만듭니다. DNB를 만들면 플로우 셀에서 라이브러리의 PCR 복사본을 생성할 필요가 없으므로 중복 읽기, 어댑터-어댑터 연결 및 PCR로 인한 오류를 상당 부분 제거할 수 있습니다.[112][citation needed]

양전하를 띤 스폿들의 패턴화된 어레이는 포토리소그래피 및 에칭 기술을 통해 제조된 후 화학적 개질을 통해 시퀀싱 플로우 셀을 생성합니다. 플로우 셀의 각 지점은 직경이 약 250nm이고 700nm(중앙에서 700nm)로 분리되어 있으며, 단일 음전하 DNB를 플로우 셀에 쉽게 부착할 수 있으므로 플로우 셀의 언더 또는 과클러스터링을 줄일 수 있습니다.[110][citation needed]
그런 다음 DNB 내의 특정 부위에 결합된 올리고뉴클레오티드 프로브를 추가하여 시퀀싱을 수행합니다. 프로브는 앵커 역할을 하여 유동 세포를 가로질러 흐르는 후 가역적으로 비활성화되고 표지된 뉴클레오티드 4개 중 하나가 결합하도록 합니다. 결합되지 않은 뉴클레오티드는 부착된 라벨의 레이저 여기 전에 세척된 다음 형광을 방출하고 신호는 염기 호출을 위한 디지털 출력으로 변환되는 카메라에 의해 캡처됩니다. 부착된 베이스에는 사이클이 완료될 때 화학적으로 절단되는 터미네이터와 라벨이 있습니다. 그 순환은 다음 뉴클레오타이드가 결합하고 그 신호가 포착되도록 하기 위해 유동 세포를 가로질러 자유로운 라벨이 붙은 뉴클레오타이드의 또 다른 흐름과 함께 반복됩니다. 이 과정은 2018년 기준 초당 약 4천만 개의 뉴클레오티드 속도로 삽입된 DNA의 서열을 결정하기 위해 여러 번(보통 50~300회) 완료됩니다.[citation needed]
솔리드 시퀀싱


Applied Biosystems(현재 라이프 테크놀로지 브랜드)의 SOLiD 기술은 결찰에 의한 시퀀싱(sequencing)을 채용하고 있습니다. 여기서, 서열화된 위치에 따라 고정된 길이의 가능한 모든 올리고뉴클레오티드 풀이 라벨링됩니다. 올리고뉴클레오티드는 어닐링되고 연결되며, 일치하는 서열에 대한 DNA 연결효소에 의한 우선적인 연결은 해당 위치의 뉴클레오티드를 정보로 하는 신호를 생성합니다. 템플릿의 각 베이스는 두 번 시퀀싱되고 결과 데이터는 이 방법에서 사용되는 2 베이스 인코딩 방식에 따라 디코딩됩니다. 시퀀싱 전에 에멀젼 PCR로 DNA를 증폭합니다. 생성된 비드는 각각 동일한 DNA 분자의 단일 사본을 포함하고 유리 슬라이드에 증착됩니다.[113] 결과는 일루미나 시퀀싱과 유사한 양과 길이의 시퀀스입니다.[77] 결찰 방법에 의한 이 시퀀싱은 회문 서열의 시퀀싱에 약간의 문제가 있는 것으로 보고되었습니다.[96]
이온 토렌트 반도체 시퀀싱
Ion Torrent Systems Inc.(현재 Life Technologies 소유)는 표준 시퀀싱 화학을 사용하지만 새로운 반도체 기반 탐지 시스템을 사용하여 시스템을 개발했습니다. 이 시퀀싱 방법은 다른 시퀀싱 시스템에서 사용되는 광학적 방법과 달리 DNA의 중합 중에 방출되는 수소 이온의 검출을 기반으로 합니다. 시퀀싱할 주형 DNA 가닥을 포함하는 마이크로웰은 단일 유형의 뉴클레오티드로 플러딩됩니다. 도입된 뉴클레오타이드가 주요 주형 뉴클레오타이드와 상보적인 경우, 그것은 성장하는 상보적 가닥에 통합됩니다. 이것은 반응이 일어났음을 나타내는 과민성 이온 센서를 유발하는 수소 이온의 방출을 유발합니다. 호모폴리머 반복이 템플릿 서열에 존재하는 경우, 다수의 뉴클레오티드가 하나의 주기로 통합됩니다. 따라서 방출된 수소의 수와 비례적으로 더 높은 전자 신호로 이어집니다.[114]

DNA 나노볼 시퀀싱
DNA 나노볼 시퀀싱은 유기체의 전체 유전체 서열을 결정하는 데 사용되는 일종의 고처리량 시퀀싱 기술입니다. Complete Genomics라는 회사는 이 기술을 사용하여 독립적인 연구자들이 제출한 샘플을 배열합니다. 이 방법은 롤링 서클 복제를 사용하여 게놈 DNA의 작은 조각을 DNA 나노볼로 증폭합니다. 결찰에 의한 비사슬 시퀀싱은 뉴클레오티드 서열을 결정하는 데 사용됩니다.[115] 이 DNA 시퀀싱 방법을 사용하면 실행당 많은 수의 DNA 나노볼을 다른 고처리량 시퀀싱 플랫폼에 비해 저렴한 시약 비용으로 시퀀싱할 수 있습니다.[116] 그러나 각 DNA 나노볼에서 DNA의 짧은 서열만 결정되므로 짧은 판독값을 참조 유전체에 매핑하는 것이 어렵습니다.[115]
헬리스코프 단일 분자 시퀀싱
헬리스코프 시퀀싱은 헬리코스 바이오사이언스가 개발한 단일 분자 시퀀싱 방법입니다. 플로우 셀 표면에 부착된 폴리-A 테일 어댑터가 추가된 DNA 단편을 사용합니다. 다음 단계는 형광 표지된 뉴클레오티드(Sanger 방법과 같이 한 번에 하나의 뉴클레오티드 유형)로 플로우 셀을 순환 세척하는 확장 기반 시퀀싱을 포함합니다. 판독은 Heliscope 시퀀서에 의해 수행됩니다.[117][118] 판독값은 평균 35bp로 짧습니다.[119] 이 기술을 특히 참신하게 만든 것은 증폭되지 않은 DNA를 염기서열 분석하여 증폭 단계와 관련된 판독 오류를 방지한 최초의 기술이라는 것입니다.[120] 2009년 헬리스코프를 사용하여 인간 게놈의 염기서열을 분석했지만 2012년 회사가 파산했습니다.[121]
미세유체시스템
DNA를 배열하는 데 사용되는 두 가지 주요 미세유체 시스템은 액적 기반 미세유체 시스템과 디지털 미세유체 시스템입니다. 미세 유체 소자는 현재 시퀀싱 어레이의 많은 현재 한계를 해결합니다.
Abate 등은 DNA 염기서열 분석을 위한 액적 기반 미세유체 장치의 사용을 연구했습니다.[4] 이 장치는 초당 수천 개의 속도로 피콜리터 크기의 물방울을 형성하고 처리할 수 있습니다. 장치는 PDMS(polydimethylsiloxane)로부터 만들어졌으며, 비말에 포함된 DNA의 서열을 판독하기 위해 FRET 분석을 사용했습니다. 배열의 각 위치는 특정 15개 염기서열에 대해 테스트되었습니다.[4]
공정한. DNA 파이로시퀀싱을 연구하기 위해 디지털 미세유체 장치를 사용했습니다.[122] 장치의 휴대성, 시약 부피, 분석 속도, 대량 제조 능력, 높은 처리량 등이 상당한 장점입니다. 이 연구는 디지털 디바이스가 파이로시퀀싱에 사용될 수 있다는 것을 보여주는 개념의 증거를 제공했습니다; 이 연구는 효소의 확장과 표지된 뉴클레오티드의 추가를 포함하는 합성을 사용하는 것을 포함했습니다.[122]
Boles et al. 는 또한 디지털 미세유체 장치에 대한 파이로시퀀싱을 연구했습니다.[123] 그들은 물방울을 만들고, 섞고, 쪼개기 위해 전기 습윤 장치를 사용했습니다. 시퀀싱은 3-효소 프로토콜과 자성 비드로 고정된 DNA 템플릿을 사용합니다. 장치는 두 가지 프로토콜을 사용하여 테스트되었으며 원시 파이로그램 수준을 기반으로 100% 정확도를 얻었습니다. 이러한 디지털 미세유체 장치의 장점은 크기, 비용 및 달성 가능한 수준의 기능 통합을 포함합니다.[123]
미세유체를 이용한 DNA 염기서열 분석 연구는 드롭스에서 방법과 같은 유사한 액적 미세유체 기술을 사용하여 RNA의 염기서열 분석에 적용할 수 있는 능력도 가지고 있습니다.[124] 이것은 이러한 많은 DNA 염기서열 분석 기술들이 유전체와 전사체에 대해 더 많이 이해하는 데 더 많이 적용되고 사용될 수 있을 것임을 보여줍니다.
개발중인 방법
현재 개발 중인 DNA 염기서열 분석 방법은 DNA 가닥이 나노포어를 통과하면서 염기서열을 판독하는 방법(현재는 상용화되어 있지만 고체 나노포어와 같은 후속 세대는 여전히 개발 중인 방법)[125][126]과 현미경 기반 기술, 육안 검출 및 기록을 위해 더 무거운 원소(예를 들어 할로겐)로 뉴클레오티드 라벨링하여 긴 DNA 조각(>5,000bp) 내에서 개별 뉴클레오티드의 위치를 식별하는 데 사용되는 원자력 현미경 또는 투과 전자 현미경과 같은 것.[127][128] 3세대 기술은 과도한 시약의 필요성을 없애고 DNA 중합효소의 공정성을 활용하여 처리량을 늘리고 결과 및 비용을 줄이는 것을 목표로 합니다.[129]
터널링 전류 DNA 염기서열 분석
또 다른 접근법은 단일 가닥 DNA가 채널을 통해 이동할 때 전기 터널링 전류 측정을 사용합니다. 전자 구조에 따라 각 베이스는 터널링 전류에 다르게 영향을 [130]주어 서로 다른 베이스 간의 차별화를 가능하게 합니다.[131]
터널링 전류의 사용은 이온 전류 방법보다 훨씬 빠른 순서로 배열할 가능성이 있으며 여러 DNA 올리고머 및 마이크로 RNA의 배열이 이미 달성되었습니다.[132]
혼성화에 의한 시퀀싱
혼성화에 의한 시퀀싱은 DNA 마이크로어레이를 사용하는 비효소적인 방법입니다. 서열이 결정될 DNA의 단일 풀은 형광 표지되고 알려진 서열을 포함하는 배열에 혼성화됩니다. 배열의 특정 지점에서 온 강력한 혼성화 신호는 염기서열 분석 중인 DNA의 염기서열을 식별합니다.[133]
이 시퀀싱 방법은 DNA 프로브라고도 불리는 짧은 단일 가닥 DNA 분자(올리고뉴클레오티드) 라이브러리의 결합 특성을 활용하여 표적 DNA 서열을 재구성합니다. 세척으로 비특이적인 잡종을 제거하고 표적 DNA를 용출합니다.[134] DNA 서열을 재구성할 수 있도록 하이브리드를 재배열합니다. 이 시퀀싱 유형의 이점은 단일 범위로 많은 수의 표적을 캡처할 수 있다는 것입니다.[135] 일반적으로 많은 화학 물질과 시작 DNA가 필요합니다. 하지만 솔루션 기반의 하이브리드화가 등장하면서 훨씬 적은 장비와 화학물질이 필요하게 되었습니다.[134]
질량 분석법을 사용한 시퀀싱
질량 분석법은 DNA 서열을 결정하는 데 사용될 수 있습니다. 특히, DNA 단편을 시각화하기 위한 겔 전기영동의 대안적인 방법으로서 매트릭스 보조 레이저 탈착 이온화 비행 시간 질량 분석법, 또는 MALDI-TOF MS가 연구되었습니다. 이 방법으로 연쇄 종결 염기 서열화 반응으로 생성된 DNA 조각을 크기가 아닌 질량으로 비교합니다. 각각의 뉴클레오티드의 질량은 다른 뉴클레오티드와 다르며 이 차이는 질량 분석법으로 감지할 수 있습니다. 단편의 단일 뉴클레오티드 돌연변이는 겔 전기영동 단독보다 MS로 더 쉽게 검출할 수 있습니다. MALDI-TOF MS는 RNA 조각 간의 차이를 보다 쉽게 감지할 수 있어 연구자들은 DNA를 먼저 RNA로 변환해 MS 기반의 방법으로 간접적으로 염기서열을 분석할 수 있습니다.[136]
MS 기반 방법에 의해 허용되는 DNA 조각의 더 높은 해상도는 법의학 연구자들에게 특별한 관심사입니다. 왜냐하면 그들은 개인을 식별하기 위해 인간 DNA 샘플에서 단일 뉴클레오티드 다형성을 발견하기를 원할 수 있기 때문입니다. 이러한 샘플은 고도로 분해될 수 있으므로 법의학 연구자들은 더 높은 안정성과 계통 연구에 적용하기 위해 미토콘드리아 DNA를 종종 선호합니다. MS 기반 염기서열 분석 방법은 연방수사국 데이터베이스에[137] 있는 샘플과 제1차 세계대전 군인들의 집단 무덤에서 발견된 뼈에서 추출한 인간 미토콘드리아 DNA의 염기서열을 비교하는 데 사용되었습니다.[138]
초기 체인 터미네이션 및 TOF MS 방법은 최대 100개 염기쌍의 읽기 길이를 보여주었습니다.[139] 연구자들은 이 평균 판독 크기를 초과할 수 없었습니다. 체인 터미네이션 시퀀싱 단독처럼 MS 기반 DNA 시퀀싱은 대규모 새로운 시퀀싱 프로젝트에 적합하지 않을 수 있습니다. 그럼에도 불구하고, 최근의 연구는 병원성 스트렙토코커스 균주의 단일 뉴클레오티드 다형성을 비교하기 위해 짧은 서열 판독과 질량 분광법을 사용했습니다.[140]
미세유체 생어 시퀀싱
미세유체 생어 시퀀싱에서는 DNA 조각의 전체 열순환 증폭과 전기영동에 의한 분리가 단일 유리 웨이퍼(직경 약 10cm)에서 이루어지므로 시약 사용량과 비용을 절감할 수 있습니다.[141] 일부 사례에서 연구자들은 마이크로칩을 사용하여 기존 시퀀싱의 처리량을 증가시킬 수 있음을 보여주었습니다.[142] 이 기술을 효과적으로 사용하기 위해서는 여전히 연구가 수행되어야 합니다.
현미경 기반 기법
이 방법은 전자현미경을 사용하여 DNA 분자의 서열을 직접 시각화합니다. 합성된 3,272개의 염기쌍 DNA 분자와 7,249개의 염기쌍 바이러스 유전체 내에서 증가된 원자수의 원자를 포함하는 변형된 염기를 효소적으로 통합하여 온전한 DNA 분자 내에서 DNA 염기쌍을 최초로 식별하는 것이 입증되었습니다.[143]
RNAP 시퀀싱
이 방법은 폴리스티렌 비드에 부착된 RNA 중합효소(RNAP)를 사용하는 것을 기반으로 합니다. 시퀀싱할 DNA의 한쪽 끝은 다른 비드에 부착되며, 양쪽 비드는 광학 트랩에 놓입니다. 전사 중 RNAP 운동은 비드를 더 가깝게 만들고 상대적인 거리 변화를 가져오며, 이는 단일 뉴클레오티드 해상도로 기록될 수 있습니다. Sanger 방법과 유사하게 4가지 뉴클레오티드 유형 각각의 농도가 낮아진 4개의 판독값을 기반으로 서열을 추론합니다.[144] 영역 간 비교를 수행하고 알려진 서열 영역과 알려지지 않은 서열 영역을 비교하여 서열 정보를 추론합니다.[145]
체외 바이러스 고처리량 시퀀싱
454 파이로시퀀싱과 시험관 내 바이러스 mRNA 디스플레이 방법의 조합을 사용하여 전체 단백질 상호작용 세트를 분석하는 방법이 개발되었습니다. 구체적으로, 이 방법은 관심 단백질을 암호화하는 mRNA에 공유적으로 연결한 다음 역전사 PCR을 사용하여 mRNA 조각을 검출합니다. 그런 다음 mRNA를 증폭하고 시퀀싱할 수 있습니다. 결합된 방법은 IVV-HiTSeq로 명명되었으며 세포가 없는 조건에서 수행할 수 있지만 결과는 생체 내 조건을 대표하지 않을 수 있습니다.[146]
DNA를 배열하는 방법은 여러 가지가 있지만 현재 시장을 지배하는 것은 소수에 불과합니다. 이에 따르면, 일루미나는 2022년 시장의 약 80%를 차지하고 있으며, 나머지 시장은 소수의 플레이어(PacBio, Oxford, 454, MGI)[147]만 차지하고 있습니다.
시료준비
DNA 시퀀싱 프로토콜의 성공은 관심 있는 생물학적 물질로부터 DNA 또는 RNA 샘플 추출 및 준비에 의존합니다.
- 성공적인 DNA 추출은 분해되지 않은 긴 가닥의 DNA 샘플을 생성합니다.
- RNA를 성공적으로 추출하면 RNA 샘플을 얻을 수 있으며, RNA 샘플은 역전사효소(역전사효소)를 사용하여 상보적 DNA(cDNA)로 변환되어야 합니다. 이는 PCR과 같은 방식으로 기존 RNA 가닥을 기반으로 상보적 DNA를 합성하는 DNA 중합효소입니다.[148] 그런 다음 상보적인 DNA를 게놈 DNA와 같은 방식으로 처리할 수 있습니다.
DNA 또는 RNA 추출 후 샘플은 시퀀싱 방법에 따라 추가 준비가 필요할 수 있습니다. Sanger 시퀀싱의 경우, 시퀀싱 전에 클로닝 절차 또는 PCR이 필요합니다. 차세대 시퀀싱 방법의 경우 처리 전 라이브러리 준비가 필요합니다.[149] 추출 후와 라이브러리 준비 후에 핵산의 품질과 양을 평가하는 것은 분해된, 단편적인, 저순도 샘플을 식별하고 고품질의 시퀀싱 데이터를 산출합니다.[150]
현재 DNA/RNA 시퀀싱 기술의 높은 처리량 특성으로 인해 샘플 준비 방법을 확장하는 데 어려움을 겪고 있습니다. 몇 가지 액체 취급 기구를 사용하여 보다 적은 수의 샘플을 준비하고 있습니다.
| 회사 | 리퀴드 핸들러 / 자동화 | lower_mark_USD | 상_마크_USD | 착지_ |
| 오픈트론 | 오픈트론 OT-2 | $6,500 | $20,000 | https://www.opentrons.com/ |
| 길손 | 길슨 피펫맥스 | $20,000 | $40,000 | https://gb.gilson.com/GBSV/system-pipetmax.html |
| 네오텍 | 네오텍 이즈메이트 | $25,000 | $45,000 | http://neotec.co.il/pipetting-device/ |
| 수식 행렬 | 포뮬러 매트릭스 맨티스 | $40,000 | $60,000 | https://formulatrix.com/liquid-handling-systems/mantis-liquid-handler/ |
| 허드슨 로보틱스 | 허드슨 로보틱스 SOLO | $40,000 | $50,000 | https://hudsonrobotics.com/products/applications/automated-solutions-next-generation-sequencing-ngs/ |
| 해밀턴 | 해밀턴 마이크로랩 님버스 | $40,000 | $80,000 | https://www.hamiltoncompany.com/automated-liquid-handling/platforms/microlab-nimbus#specifications |
| TTP 랩테크 | TTP 랩테크 모기 HV 유전체학 | $45,000 | $80,000 | https://www.sptlabtech.com/products/liquid-handling/mosquito-hv-genomics/ |
| 벡맨 콜터 | 바이오멕 4000 | $50,000 | $65,000 | https://www.mybeckman.uk/liquid-handlers/biomek-4000/b22640 |
| 해밀턴 | 해밀턴 게놈 스타렛 | $50,000 | $100,000 | https://www.hamiltoncompany.com/automated-liquid-handling/assay-ready-workstations/genomic-starlet |
| 에펜도르프 | 에펜도르프 ep모션 5075t | $95,000 | $110,000 | https://www.eppendorf.com/epmotion/ |
| 벡맨 콜터 | 벡맨 콜터 바이오메키5 | $100,000 | $150,000 | https://www.beckman.com/liquid-handlers/biomek-i5 |
| 해밀턴 | 해밀턴 NGS 별 | $100,000 | $200,000 | http://www.hamiltonrobotics.com/ |
| 퍼킨엘머 | 퍼킨엘머 스크론 G3 NGS 및 NGSx 워크스테이션 | $150,000 | $220,000 | https://www.perkinelmer.com/uk/product/sciclone-g3-ngs-workstation-cls145321 |
| 어질런트 | 애질런트 브라보 NGS | $170,000 | $290,000 | https://www.agilent.com/en/products/automated-liquid-handling/automated-liquid-handling-applications/bravo-ngs |
| 벡맨 콜터 | 벡맨 콜터 바이오메키7 | $200,000 | $250,000 | https://www.beckman.com/liquid-handlers/biomek-i7 |
| 랩시테 에코 525 | 벡맨 콜터 랩시테 에코 525 | $260,000 | $300,000 | https://www.labcyte.com/products/liquid-handling/echo-525-liquid-handler |
| 테칸 | 테칸 NGS | $270,000 | $350,000 | https://lifesciences.tecan.com/ngs-sample-preparation |
개발 이니셔티브

2006년 10월, X Prize Foundation은 Archon X Prize라고 불리는 완전한 게놈 시퀀싱 기술의 개발을 촉진하기 위한 계획을 세웠고, "10일 이내에 장치를 만들어 100개의 인간 게놈을 시퀀싱하는 데 사용할 수 있는 최초의 팀"에 1,000만 달러를 수여할 계획이었습니다. 염기서열 분석을 할 때마다 오류가 1개 이하로 정확하게 유전체의 98% 이상을 차지하며, 유전체당 1만 달러 이하의 반복적인 비용으로 염기서열 분석을 수행합니다."[151]
매년 국립 인간 유전체 연구소, 즉 NHGRI는 유전체학의 새로운 연구와 개발을 위한 보조금을 장려합니다. 2010년 보조금 및 2011년 후보에는 미세유체, 폴리니 및 염기성 염기서열 분석 방법론에 대한 지속적인 연구가 포함되어 있습니다.[152]
계산상의 어려움
여기에 설명된 시퀀싱 기술은 완전한 유전체(서열 조립)와 같은 더 긴 서열로 조립되어야 하는 원시 데이터를 생성합니다. 이를 달성하기 위해서는 Pred 및 Phrap과 같은 프로그램 및 알고리즘에 의해 수행되는 원시 시퀀스 데이터의 평가와 같은 많은 계산 문제가 있습니다. 다른 문제들은 유전체의 여러 곳에서 발생하기 때문에 종종 완전한 유전체 집합체를 막는 반복적인 서열을 다루어야 합니다. 그 결과 많은 수의 염기서열이 특정 염색체에 할당되지 않을 수 있습니다. 원시 서열 데이터의 생산은 상세한 생물정보학적 분석의 시작에 불과합니다.[153] 그러나 서열화 오류를 수정하고 서열화하는 새로운 방법이 개발되었습니다.[154]
트리밍 읽기
때때로 시퀀서에서 생성된 원시 판독값은 길이의 일부에 대해서만 정확하고 정확합니다. 전체 읽기를 사용하면 유전체 어셈블리, SNP 호출 또는 유전자 발현 추정과 같은 다운스트림 분석에 인공물을 도입할 수 있습니다. 알고리즘의 윈도우 기반 또는 실행-합 클래스를 기반으로 두 가지 클래스의 트리밍 프로그램이 도입되었습니다.[155] 다음은 현재 사용 가능한 트리밍 알고리즘의 일부 목록이며 해당 알고리즘 클래스를 지정합니다.
| 알고리즘명 | 알고리즘의 종류 | 링크 |
|---|---|---|
| 컷어댑트[156] | 주행합 | 컷어댑트 |
| 콘데트리[157] | 창 기반 | 콘데트리 |
| ERN-Filter[158] | 주행합 | ERN-Filter |
| FASTX품질트리머 | 창 기반 | FASTX품질트리머 |
| 프린세스큐[159] | 창 기반 | 프린세스큐 |
| 트리모마틱[160] | 창 기반 | 트리모마틱 |
| 솔렉사QA[161] | 창 기반 | 솔렉사QA |
| SolexaQA-BWA | 주행합 | SolexaQA-BWA |
| 낫 | 창 기반 | 낫 |
윤리적 문제
이 구간은 확장이 필요합니다. 추가하여 도울 수 있습니다. (2015년 5월) |
인간 유전학은 1970년대[162] 초부터 생명윤리학 분야에 포함되어 왔으며 DNA 염기서열 분석(특히 고처리량 염기서열 분석) 사용의 성장은 많은 윤리적 문제를 초래했습니다. 한 가지 중요한 문제는 개인의 DNA와 그 DNA가 염기서열 분석될 때 생성되는 데이터의 소유권입니다.[163] DNA 분자 자체와 관련하여, 이 주제에 대한 주요 법률 사건인 캘리포니아 대학(1990)의 Moore v. Regents는 개인은 버려지는 세포에 대한 재산권 또는 이 세포를 사용하여 얻은 이익(예를 들어, 특허 세포주로서)을 갖지 않는다고 판결했습니다. 단, 개인은 세포의 제거 및 사용에 대해 동의할 권리가 있습니다. DNA 염기서열 분석을 통해 생성된 데이터와 관련하여 무어는 개인에게 자신의 DNA에서 파생된 정보에 대한 권리를 부여하지 않습니다.[163]
DNA 염기서열 분석이 널리 퍼지면서 유전체 데이터의 저장, 보안, 공유도 중요해졌습니다.[163][164] 예를 들어, 보험사들이 개인의 DNA에 기초한 개인의 인지된 미래 건강에 따라 개인의 게놈 데이터를 사용하여 견적을 수정할 수 있다는 우려가 있습니다.[164][165] 2008년 5월 미국에서는 유전자 정보 차별금지법(GINA)이 체결되어 건강보험 및 고용과 관련하여 유전자 정보를 근거로 차별하는 것을 금지하고 있습니다.[166][167] 2012년 미국 대통령 생명윤리문제연구위원회는 GINA와 같은 DNA 염기서열결정 데이터에 대한 기존의 개인정보 보호법과 건강보험의 휴대성 및 책임법이 불충분하다고 보고 전체 유전체 염기서열결정 데이터가 특히 민감하다고 지적했습니다. 데이터가 생성된 개인뿐만 아니라 그들의 친척을 식별하는 데 사용될 수 있기 때문입니다.[168][169]
대부분의 미국에서, 핥은 우표나 봉투, 커피 컵, 담배, 껌, 가정 쓰레기, 공공 보도에 떨어진 머리카락에서 발견된 것과 같은 "버려진" DNA는 경찰, 개인 수사관, 정치적 반대자를 포함하여 누구나 합법적으로 수집하고 시퀀싱 할 수 있습니다. 또는 친자 관계 분쟁에 연루된 사람들. 2013년 현재 11개 주에서 "DNA 도용"을 금지하는 것으로 해석할 수 있는 법률이 있습니다.[170]
23과 Me와 같은 회사에서 신생아와 성인 모두에서 유전자 변이 검사의 사용이 증가함에 따라 윤리적 문제도 제기되었습니다.[171][172] 유전자 변이를 선별하는 것이 해로울 수 있다는 주장이 제기돼 질병 위험이 높아진 것으로 밝혀진 개인들의 불안감이 커지고 있습니다.[173] 예를 들어, 타임지에 언급된 한 사례에서, 유전적 변이에 대해 아픈 아기를 선별하는 의사들은 치매가 부모에게 야기할 수 있는 해악 때문에 치매와 관련된 관련 없는 변이를 부모에게 알리지 않기로 선택했습니다.[174] 하지만, 2011년 뉴잉글랜드 의학 저널의 한 연구는 질병 위험 프로파일링을 받는 사람들이 증가된 수준의 불안을 보여주지 않았다는 것을 보여주었습니다.[173] 또한 나노포어 기반 염기서열 분석과 같은 차세대 염기서열 분석 기술의 발전으로 윤리적인 문제도 더욱 제기되고 있습니다.[175]
참고 항목
- 생물정보학 – 대규모의 복잡한 생물학적 데이터 세트의 전산 분석
- 암유전체 염기서열 분석
- 순환정합순서
- DNA 컴퓨팅 – 분자생물학 하드웨어를 이용한 컴퓨팅
- DNA 전계 효과 트랜지스터 – 하는 DNA 의 부분 전하로 인한 전계 효과를 사용하는 트랜지스터
- DNA 염기서열결정이론 – 생물학적 이론
- DNA 시퀀서 – DNA 시퀀싱 프로세스를 자동화하는 데 사용되는 과학 기기
- 지노그래픽 프로젝트 – 시민 과학 프로젝트
- 게놈 프로젝트 – 하는 프로젝트 페이지 유형
- 멸종위기종의 유전체 염기서열 분석 – 멸종위기 평가를 위한 DNA 검사
- 게놈 스키밍 – 게놈 시퀀싱 방법
- IsoBase – PPI 네트워크 전반에 걸쳐 기능적으로 관련된 단백질 하는 페이지
- 링크드-리드 시퀀싱
- 점프 라이브러리
- 핵산 서열 – 핵산 내 뉴클레오티드의 연속
- 다중 결찰 의존 프로브 증폭
- 개인 맞춤형 의료 – 개별 환자에게 의료 행위를 맞춤형으로 제공하는 의료 모델
- 단백질 서열 분석 – 단백질의 아미노산 배열 분석
- 시퀀스 마이닝
- 시퀀스 프로파일링 도구
- 혼성화에 의한 염기서열 분석 – 을 폴백( 가닥에서 고정된 크기의 구성 뉴클레오티드를 결정하는 방법
- 결찰에 의한 시퀀싱 – 효소 DNA 연결효소를 사용하여 DNA 서열에서 특정 위치에 존재하는 뉴클레오티드를 식별하는 DNA 서열화 방법위키데이터 하는 페이지
- TIARA(데이터베이스) – 개인 유전체 정보 데이터베이스
- 투과전자현미경 DNA 염기서열 분석 – 단분자 염기서열 분석 기술
메모들
- ^ "차세대"는 2019년 현재 광범위하게 사용되고 있습니다. 예를 들어. Straiton J, Free T, Sawyer A, Martin J (February 2019). "From Sanger Sequencing to Genome Databases and Beyond". BioTechniques. 66 (2): 60–63. doi:10.2144/btn-2019-0011. PMID 30744413.
Next-generation sequencing (NGS) technologies have revolutionized genomic research. (opening sentence of the article)
참고문헌
- ^ "Introducing 'dark DNA' – the phenomenon that could change how we think about evolution". 24 August 2017.
- ^ Behjati S, Tarpey PS (December 2013). "What is next generation sequencing?". Archives of Disease in Childhood: Education and Practice Edition. 98 (6): 236–8. doi:10.1136/archdischild-2013-304340. PMC 3841808. PMID 23986538.
- ^ Chmielecki J, Meyerson M (14 January 2014). "DNA sequencing of cancer: what have we learned?". Annual Review of Medicine. 65 (1): 63–79. doi:10.1146/annurev-med-060712-200152. PMID 24274178.
- ^ a b c d Abate AR, Hung T, Sperling RA, Mary P, Rotem A, Agresti JJ, et al. (December 2013). "DNA sequence analysis with droplet-based microfluidics". Lab on a Chip. 13 (24): 4864–9. doi:10.1039/c3lc50905b. PMC 4090915. PMID 24185402.
- ^ Pekin D, Skhiri Y, Baret JC, Le Corre D, Mazutis L, Salem CB, et al. (July 2011). "Quantitative and sensitive detection of rare mutations using droplet-based microfluidics". Lab on a Chip. 11 (13): 2156–66. doi:10.1039/c1lc20128j. PMID 21594292.
- ^ Olsvik O, Wahlberg J, Petterson B, Uhlén M, Popovic T, Wachsmuth IK, Fields PI (January 1993). "Use of automated sequencing of polymerase chain reaction-generated amplicons to identify three types of cholera toxin subunit B in Vibrio cholerae O1 strains". J. Clin. Microbiol. 31 (1): 22–25. doi:10.1128/JCM.31.1.22-25.1993. PMC 262614. PMID 7678018.

- ^ Pettersson E, Lundeberg J, Ahmadian A (February 2009). "Generations of sequencing technologies". Genomics. 93 (2): 105–11. doi:10.1016/j.ygeno.2008.10.003. PMID 18992322.
- ^ a b c Jay E, Bambara R, Padmanabhan R, Wu R (March 1974). "DNA sequence analysis: a general, simple and rapid method for sequencing large oligodeoxyribonucleotide fragments by mapping". Nucleic Acids Research. 1 (3): 331–53. doi:10.1093/nar/1.3.331. PMC 344020. PMID 10793670.
- ^ Hunt, Katie (17 February 2021). "World's oldest DNA sequenced from a mammoth that lived more than a million years ago". CNN. Retrieved 17 February 2021.
- ^ Callaway, Ewen (17 February 2021). "Million-year-old mammoth genomes shatter record for oldest ancient DNA – Permafrost-preserved teeth, up to 1.6 million years old, identify a new kind of mammoth in Siberia". Nature. 590 (7847): 537–538. Bibcode:2021Natur.590..537C. doi:10.1038/d41586-021-00436-x. PMID 33597786.
- ^ a b c Castro, Christina; Marine, Rachel; Ramos, Edward; Ng, Terry Fei Fan (2019). "The effect of variant interference on de novo assembly for viral deep sequencing". BMC Genomics. 21 (1): 421. bioRxiv 10.1101/815480. doi:10.1186/s12864-020-06801-w. PMC 7306937. PMID 32571214.
- ^ a b Wohl, Shirlee; Schaffner, Stephen F.; Sabeti, Pardis C. (2016). "Genomic Analysis of Viral Outbreaks". Annual Review of Virology. 3 (1): 173–195. doi:10.1146/annurev-virology-110615-035747. PMC 5210220. PMID 27501264.
- ^ Boycott, Kym M.; Vanstone, Megan R.; Bulman, Dennis E.; MacKenzie, Alex E. (October 2013). "Rare-disease genetics in the era of next-generation sequencing: discovery to translation". Nature Reviews Genetics. 14 (10): 681–691. doi:10.1038/nrg3555. PMID 23999272. S2CID 8496181.
- ^ Schleusener V, Köser CU, Beckert P, Niemann S, Feuerriegel S (2017). "Mycobacterium tuberculosis resistance prediction and lineage classification from genome sequencing: comparison of automated analysis tools". Sci Rep. 7: 46327. Bibcode:2017NatSR...746327S. doi:10.1038/srep46327. PMC 7365310. PMID 28425484.
- ^ Mahé P, El Azami M, Barlas P, Tournoud M (2019). "A large scale evaluation of TBProfiler and Mykrobe for antibiotic resistance prediction in Mycobacterium tuberculosis". PeerJ. 7: e6857. doi:10.7717/peerj.6857. PMC 6500375. PMID 31106066.
- ^ Mykrove predictor – 전체 유전체 서열 데이터로부터 황색포도상구균 및 M. tuberculosis에 대한 항생제 내성 예측
- ^ Bradley, Phelim; Gordon, N. Claire; Walker, Timothy M.; Dunn, Laura; Heys, Simon; Huang, Bill; Earle, Sarah; Pankhurst, Louise J.; Anson, Luke; de Cesare, Mariateresa; Piazza, Paolo; Votintseva, Antonina A.; Golubchik, Tanya; Wilson, Daniel J.; Wyllie, David H.; Diel, Roland; Niemann, Stefan; Feuerriegel, Silke; Kohl, Thomas A.; Ismail, Nazir; Omar, Shaheed V.; Smith, E. Grace; Buck, David; McVean, Gil; Walker, A. Sarah; Peto, Tim E. A.; Crook, Derrick W.; Iqbal, Zamin (21 December 2015). "Rapid antibiotic-resistance predictions from genome sequence data for Staphylococcus aureus and Mycobacterium tuberculosis". Nature Communications. 6 (1): 10063. Bibcode:2015NatCo...610063B. doi:10.1038/ncomms10063. PMC 4703848. PMID 26686880.
- ^ "Michael Mosley vs the superbugs". Archived from the original on 24 November 2020. Retrieved 21 October 2019.
- ^ Mykrobe, Mykrobe-tools, 24 December 2022, retrieved 2 January 2023
- ^ Curtis C, Hereward J (29 August 2017). "From the crime scene to the courtroom: the journey of a DNA sample". The Conversation.
- ^ Moréra S, Larivière L, Kurzeck J, Aschke-Sonnenborn U, Freemont PS, Janin J, Rüger W (August 2001). "High resolution crystal structures of T4 phage beta-glucosyltransferase: induced fit and effect of substrate and metal binding". Journal of Molecular Biology. 311 (3): 569–77. doi:10.1006/jmbi.2001.4905. PMID 11493010.
- ^ Ehrlich M, Gama-Sosa MA, Huang LH, Midgett RM, Kuo KC, McCune RA, Gehrke C (April 1982). "Amount and distribution of 5-methylcytosine in human DNA from different types of tissues of cells". Nucleic Acids Research. 10 (8): 2709–21. doi:10.1093/nar/10.8.2709. PMC 320645. PMID 7079182.
- ^ Ehrlich M, Wang RY (June 1981). "5-Methylcytosine in eukaryotic DNA". Science. 212 (4501): 1350–7. Bibcode:1981Sci...212.1350E. doi:10.1126/science.6262918. PMID 6262918.
- ^ Song CX, Clark TA, Lu XY, Kislyuk A, Dai Q, Turner SW, et al. (November 2011). "Sensitive and specific single-molecule sequencing of 5-hydroxymethylcytosine". Nature Methods. 9 (1): 75–7. doi:10.1038/nmeth.1779. PMC 3646335. PMID 22101853.
- ^ Czernecki, Dariusz; Bonhomme, Frédéric; Kaminski, Pierre-Alexandre; Delarue, Marc (5 August 2021). "Characterization of a triad of genes in cyanophage S-2L sufficient to replace adenine by 2-aminoadenine in bacterial DNA". Nature Communications. 12 (1): 4710. Bibcode:2021NatCo..12.4710C. doi:10.1038/s41467-021-25064-x. PMC 8342488. PMID 34354070. S2CID 233745192.
- ^ Watson JD, Crick FH (1953). "The structure of DNA". Cold Spring Harb. Symp. Quant. Biol. 18: 123–31. doi:10.1101/SQB.1953.018.01.020. PMID 13168976.
- ^ Marks, L. "The path to DNA sequencing: The life and work of Frederick Sanger". What is Biotechnology?. Retrieved 27 June 2023.
- ^ Min Jou W, Haegeman G, Ysebaert M, Fiers W (May 1972). "Nucleotide sequence of the gene coding for the bacteriophage MS2 coat protein". Nature. 237 (5350): 82–8. Bibcode:1972Natur.237...82J. doi:10.1038/237082a0. PMID 4555447. S2CID 4153893.
- ^ Fiers W, Contreras R, Duerinck F, Haegeman G, Iserentant D, Merregaert J, Min Jou W, Molemans F, Raeymaekers A, Van den Berghe A, Volckaert G, Ysebaert M (April 1976). "Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene". Nature. 260 (5551): 500–7. Bibcode:1976Natur.260..500F. doi:10.1038/260500a0. PMID 1264203. S2CID 4289674.
- ^ Ozsolak F, Milos PM (February 2011). "RNA sequencing: advances, challenges and opportunities". Nature Reviews Genetics. 12 (2): 87–98. doi:10.1038/nrg2934. PMC 3031867. PMID 21191423.
- ^ "Ray Wu Faculty Profile". Cornell University. Archived from the original on 4 March 2009.
- ^ Padmanabhan R, Jay E, Wu R (June 1974). "Chemical synthesis of a primer and its use in the sequence analysis of the lysozyme gene of bacteriophage T4". Proceedings of the National Academy of Sciences of the United States of America. 71 (6): 2510–4. Bibcode:1974PNAS...71.2510P. doi:10.1073/pnas.71.6.2510. PMC 388489. PMID 4526223.
- ^ Onaga LA (June 2014). "Ray Wu as Fifth Business: Demonstrating Collective Memory in the History of DNA Sequencing". Studies in the History and Philosophy of Science. Part C. 46: 1–14. doi:10.1016/j.shpsc.2013.12.006. PMID 24565976.
- ^ Wu R (1972). "Nucleotide sequence analysis of DNA". Nature New Biology. 236 (68): 198–200. doi:10.1038/newbio236198a0. PMID 4553110.
- ^ Padmanabhan R, Wu R (1972). "Nucleotide sequence analysis of DNA. IX. Use of oligonucleotides of defined sequence as primers in DNA sequence analysis". Biochem. Biophys. Res. Commun. 48 (5): 1295–302. doi:10.1016/0006-291X(72)90852-2. PMID 4560009.
- ^ Wu R, Tu CD, Padmanabhan R (1973). "Nucleotide sequence analysis of DNA. XII. The chemical synthesis and sequence analysis of a dodecadeoxynucleotide which binds to the endolysin gene of bacteriophage lambda". Biochem. Biophys. Res. Commun. 55 (4): 1092–99. doi:10.1016/S0006-291X(73)80007-5. PMID 4358929.
- ^ a b Sanger F, Nicklen S, Coulson AR (December 1977). "DNA sequencing with chain-terminating inhibitors". Proc. Natl. Acad. Sci. USA. 74 (12): 5463–77. Bibcode:1977PNAS...74.5463S. doi:10.1073/pnas.74.12.5463. PMC 431765. PMID 271968.
- ^ a b c Maxam AM, Gilbert W (February 1977). "A new method for sequencing DNA". Proc. Natl. Acad. Sci. USA. 74 (2): 560–64. Bibcode:1977PNAS...74..560M. doi:10.1073/pnas.74.2.560. PMC 392330. PMID 265521.
- ^ 길버트, W.D. DNA 염기서열 분석과 유전자 구조. 1980년 12월 8일 노벨 강연.
- ^ Gilbert W, Maxam A (December 1973). "The Nucleotide Sequence of the lac Operator". Proc. Natl. Acad. Sci. U.S.A. 70 (12): 3581–84. Bibcode:1973PNAS...70.3581G. doi:10.1073/pnas.70.12.3581. PMC 427284. PMID 4587255.
- ^ Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, Hutchison CA, Slocombe PM, Smith M (February 1977). "Nucleotide sequence of bacteriophage phi X174 DNA". Nature. 265 (5596): 687–95. Bibcode:1977Natur.265..687S. doi:10.1038/265687a0. PMID 870828. S2CID 4206886.
- ^ Marks, L. "The next frontier: Human viruses". What is Biotechnology?. Retrieved 27 June 2023.
- ^ Beck S, Pohl FM (1984). "DNA sequencing with direct blotting electrophoresis". EMBO J. 3 (12): 2905–09. doi:10.1002/j.1460-2075.1984.tb02230.x. PMC 557787. PMID 6396083.
- ^ 미국 특허 4,631,122 (1986)
- ^ Feldmann H, et al. (1994). "Complete DNA sequence of yeast chromosome II". EMBO J. 13 (24): 5795–809. doi:10.1002/j.1460-2075.1994.tb06923.x. PMC 395553. PMID 7813418.
- ^ Smith LM, Sanders JZ, Kaiser RJ, Hughes P, Dodd C, Connell CR, Heiner C, Kent SB, Hood LE (12 June 1986). "Fluorescence Detection in Automated DNA Sequence Analysis". Nature. 321 (6071): 674–79. Bibcode:1986Natur.321..674S. doi:10.1038/321674a0. PMID 3713851. S2CID 27800972.
- ^ Prober JM, Trainor GL, Dam RJ, Hobbs FW, Robertson CW, Zagursky RJ, Cocuzza AJ, Jensen MA, Baumeister K (16 October 1987). "A system for rapid DNA sequencing with fluorescent chain-terminating dideoxynucleotides". Science. 238 (4825): 336–41. Bibcode:1987Sci...238..336P. doi:10.1126/science.2443975. PMID 2443975.
- ^ Adams MD, Kelley JM, Gocayne JD, Dubnick M, Polymeropoulos MH, Xiao H, Merril CR, Wu A, Olde B, Moreno RF (June 1991). "Complementary DNA sequencing: expressed sequence tags and human genome project". Science. 252 (5013): 1651–56. Bibcode:1991Sci...252.1651A. doi:10.1126/science.2047873. PMID 2047873. S2CID 13436211.
- ^ Fleischmann RD, Adams MD, White O, Clayton RA, Kirkness EF, Kerlavage AR, Bult CJ, Tomb JF, Dougherty BA, Merrick JM (July 1995). "Whole-genome random sequencing and assembly of Haemophilus influenzae Rd". Science. 269 (5223): 496–512. Bibcode:1995Sci...269..496F. doi:10.1126/science.7542800. PMID 7542800.
- ^ Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, et al. (February 2001). "Initial sequencing and analysis of the human genome" (PDF). Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Venter JC, Adams MD, et al. (February 2001). "The sequence of the human genome". Science. 291 (5507): 1304–51. Bibcode:2001Sci...291.1304V. doi:10.1126/science.1058040. PMID 11181995.
- ^ Yang, Aimin; Zhang, Wei; Wang, Jiahao; Yang, Ke; Han, Yang; Zhang, Limin (2020). "Review on the Application of Machine Learning Algorithms in the Sequence Data Mining of DNA". Frontiers in Bioengineering and Biotechnology. 8: 1032. doi:10.3389/fbioe.2020.01032. PMC 7498545. PMID 33015010.
- ^ "Espacenet – Bibliographic data". worldwide.espacenet.com.
- ^ Ronaghi M, Karamohamed S, Pettersson B, Uhlén M, Nyrén P (1996). "Real-time DNA sequencing using detection of pyrophosphate release". Analytical Biochemistry. 242 (1): 84–89. doi:10.1006/abio.1996.0432. PMID 8923969.
- ^ a b Kawashima, Eric H.; Laurent Farinelli; Pascal Mayer (12 May 2005). "Patent: Method of nucleic acid amplification". Archived from the original on 22 February 2013. Retrieved 22 December 2012.
- ^ Ewing B, Green P (March 1998). "Base-calling of automated sequencer traces using phred. II. Error probabilities". Genome Res. 8 (3): 186–94. doi:10.1101/gr.8.3.186. PMID 9521922.
- ^ "Quality Scores for Next-Generation Sequencing" (PDF). Illumina. 31 October 2011. Retrieved 8 May 2018.
- ^ a b Brenner S, Johnson M, Bridgham J, Golda G, Lloyd DH, Johnson D, Luo S, McCurdy S, Foy M, Ewan M, Roth R, George D, Eletr S, Albrecht G, Vermaas E, Williams SR, Moon K, Burcham T, Pallas M, DuBridge RB, Kirchner J, Fearon K, Mao J, Corcoran K (2000). "Gene expression analysis by massively parallel signature sequencing (MPSS) on microbead arrays". Nature Biotechnology. 18 (6): 630–34. doi:10.1038/76469. PMID 10835600. S2CID 13884154.
- ^ "maxam gilbert sequencing". PubMed.
- ^ Sanger F, Coulson AR (May 1975). "A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase". J. Mol. Biol. 94 (3): 441–48. doi:10.1016/0022-2836(75)90213-2. PMID 1100841.
- ^ Wetterstrand, Kris. "DNA Sequencing Costs: Data from the NHGRI Genome Sequencing Program (GSP)". National Human Genome Research Institute. Retrieved 30 May 2013.
- ^ Nyren, P.; Pettersson, B.; Uhlen, M. (January 1993). "Solid Phase DNA Minisequencing by an Enzymatic Luminometric Inorganic Pyrophosphate Detection Assay". Analytical Biochemistry. 208 (1): 171–175. doi:10.1006/abio.1993.1024. PMID 8382019.
- ^ Ronaghi, Mostafa; Uhlén, Mathias; Nyrén, Pål (17 July 1998). "A Sequencing Method Based on Real-Time Pyrophosphate". Science. 281 (5375): 363–365. doi:10.1126/science.281.5375.363. PMID 9705713. S2CID 26331871.
- ^ Quail MA, Gu Y, Swerdlow H, Mayho M (2012). "Evaluation and optimisation of preparative semi-automated electrophoresis systems for Illumina library preparation". Electrophoresis. 33 (23): 3521–28. doi:10.1002/elps.201200128. PMID 23147856. S2CID 39818212.
- ^ Duhaime MB, Deng L, Poulos BT, Sullivan MB (2012). "Towards quantitative metagenomics of wild viruses and other ultra-low concentration DNA samples: a rigorous assessment and optimization of the linker amplification method". Environ. Microbiol. 14 (9): 2526–37. Bibcode:2012EnvMi..14.2526D. doi:10.1111/j.1462-2920.2012.02791.x. PMC 3466414. PMID 22713159.
- ^ Peterson BK, Weber JN, Kay EH, Fisher HS, Hoekstra HE (2012). "Double digest RADseq: an inexpensive method for de novo SNP discovery and genotyping in model and non-model species". PLOS ONE. 7 (5): e37135. Bibcode:2012PLoSO...737135P. doi:10.1371/journal.pone.0037135. PMC 3365034. PMID 22675423.
- ^ Williams R, Peisajovich SG, Miller OJ, Magdassi S, Tawfik DS, Griffiths AD (2006). "Amplification of complex gene libraries by emulsion PCR". Nature Methods. 3 (7): 545–50. doi:10.1038/nmeth896. PMID 16791213. S2CID 27459628.
- ^ a b Margulies M, Egholm M, et al. (September 2005). "Genome Sequencing in Open Microfabricated High Density Picoliter Reactors". Nature. 437 (7057): 376–80. Bibcode:2005Natur.437..376M. doi:10.1038/nature03959. PMC 1464427. PMID 16056220.
- ^ Shendure J, Porreca GJ, Reppas NB, Lin X, McCutcheon JP, Rosenbaum AM, Wang MD, Zhang K, Mitra RD, Church GM (2005). "Accurate Multiplex Polony Sequencing of an Evolved Bacterial Genome". Science. 309 (5741): 1728–32. Bibcode:2005Sci...309.1728S. doi:10.1126/science.1117389. PMID 16081699. S2CID 11405973.
- ^ "Applied Biosystems – File Not Found (404 Error)". 16 May 2008. Archived from the original on 16 May 2008.
- ^ Goodwin S, McPherson JD, McCombie WR (May 2016). "Coming of age: ten years of next-generation sequencing technologies". Nature Reviews Genetics. 17 (6): 333–51. doi:10.1038/nrg.2016.49. PMC 10373632. PMID 27184599. S2CID 8295541.
- ^ Staden R (11 June 1979). "A strategy of DNA sequencing employing computer programs". Nucleic Acids Research. 6 (7): 2601–10. doi:10.1093/nar/6.7.2601. PMC 327874. PMID 461197.
- ^ de Magalhães JP, Finch CE, Janssens G (2010). "Next-generation sequencing in aging research: emerging applications, problems, pitfalls and possible solutions". Ageing Research Reviews. 9 (3): 315–23. doi:10.1016/j.arr.2009.10.006. PMC 2878865. PMID 19900591.
- ^ Grada A (August 2013). "Next-generation sequencing: methodology and application". J Invest Dermatol. 133 (8): e11. doi:10.1038/jid.2013.248. PMID 23856935.
- ^ Hall N (May 2007). "Advanced sequencing technologies and their wider impact in microbiology". J. Exp. Biol. 210 (Pt 9): 1518–25. doi:10.1242/jeb.001370. PMID 17449817.

- ^ Church GM (January 2006). "Genomes for all". Sci. Am. 294 (1): 46–54. Bibcode:2006SciAm.294a..46C. doi:10.1038/scientificamerican0106-46. PMID 16468433. S2CID 28769137.(구독 필수)
- ^ a b c Schuster SC (January 2008). "Next-generation sequencing transforms today's biology". Nat. Methods. 5 (1): 16–18. doi:10.1038/nmeth1156. PMID 18165802. S2CID 1465786.
- ^ Kalb, Gilbert; Moxley, Robert (1992). Massively Parallel, Optical, and Neural Computing in the United States. IOS Press. ISBN 978-90-5199-097-3.[페이지 필요]
- ^ ten Bosch JR, Grody WW (2008). "Keeping Up with the Next Generation". The Journal of Molecular Diagnostics. 10 (6): 484–92. doi:10.2353/jmoldx.2008.080027. PMC 2570630. PMID 18832462.

- ^ Tucker T, Marra M, Friedman JM (2009). "Massively Parallel Sequencing: The Next Big Thing in Genetic Medicine". The American Journal of Human Genetics. 85 (2): 142–54. doi:10.1016/j.ajhg.2009.06.022. PMC 2725244. PMID 19679224.

- ^ a b Straiton J, Free T, Sawyer A, Martin J (February 2019). "From Sanger sequencing to genome databases and beyond". BioTechniques. Future Science. 66 (2): 60–63. doi:10.2144/btn-2019-0011. PMID 30744413.
- ^ Quail MA, Smith M, Coupland P, Otto TD, Harris SR, Connor TR, Bertoni A, Swerdlow HP, Gu Y (1 January 2012). "A tale of three next generation sequencing platforms: comparison of Ion Torrent, Pacific Biosciences and illumina MiSeq sequencers". BMC Genomics. 13 (1): 341. doi:10.1186/1471-2164-13-341. PMC 3431227. PMID 22827831.

- ^ Liu L, Li Y, Li S, Hu N, He Y, Pong R, Lin D, Lu L, Law M (1 January 2012). "Comparison of Next-Generation Sequencing Systems". Journal of Biomedicine and Biotechnology. 2012: 251364. doi:10.1155/2012/251364. PMC 3398667. PMID 22829749.

- ^ a b c "New Software, Polymerase for Sequel System Boost Throughput and Affordability – PacBio". 7 March 2018.
- ^ "After a Year of Testing, Two Early PacBio Customers Expect More Routine Use of RS Sequencer in 2012". GenomeWeb. 10 January 2012.(등록필요)
- ^ Inc., Pacific Biosciences (2013). "Pacific Biosciences Introduces New Chemistry With Longer Read Lengths to Detect Novel Features in DNA Sequence and Advance Genome Studies of Large Organisms" (Press release).
{{cite press release}}:last=일반 이름(도움말)이 있습니다. - ^ Chin CS, Alexander DH, Marks P, Klammer AA, Drake J, Heiner C, Clum A, Copeland A, Huddleston J, Eichler EE, Turner SW, Korlach J (2013). "Nonhybrid, finished microbial genome assemblies from long-read SMRT sequencing data". Nat. Methods. 10 (6): 563–69. doi:10.1038/nmeth.2474. PMID 23644548. S2CID 205421576.
- ^ a b "De novo bacterial genome assembly: a solved problem?". 5 July 2013.
- ^ Rasko DA, Webster DR, Sahl JW, Bashir A, Boisen N, Scheutz F, Paxinos EE, Sebra R, Chin CS, Iliopoulos D, Klammer A, Peluso P, Lee L, Kislyuk AO, Bullard J, Kasarskis A, Wang S, Eid J, Rank D, Redman JC, Steyert SR, Frimodt-Møller J, Struve C, Petersen AM, Krogfelt KA, Nataro JP, Schadt EE, Waldor MK (25 August 2011). "Origins of the Strain Causing an Outbreak of Hemolytic–Uremic Syndrome in Germany". N Engl J Med. 365 (8): 709–17. doi:10.1056/NEJMoa1106920. PMC 3168948. PMID 21793740.

- ^ Tran B, Brown AM, Bedard PL, Winquist E, Goss GD, Hotte SJ, Welch SA, Hirte HW, Zhang T, Stein LD, Ferretti V, Watt S, Jiao W, Ng K, Ghai S, Shaw P, Petrocelli T, Hudson TJ, Neel BG, Onetto N, Siu LL, McPherson JD, Kamel-Reid S, Dancey JE (1 January 2012). "Feasibility of real time next generation sequencing of cancer genes linked to drug response: Results from a clinical trial". Int. J. Cancer. 132 (7): 1547–55. doi:10.1002/ijc.27817. PMID 22948899. S2CID 72705.(구독 필수)
- ^ Murray IA, Clark TA, Morgan RD, Boitano M, Anton BP, Luong K, Fomenkov A, Turner SW, Korlach J, Roberts RJ (2 October 2012). "The methylomes of six bacteria". Nucleic Acids Research. 40 (22): 11450–62. doi:10.1093/nar/gks891. PMC 3526280. PMID 23034806.
- ^ "Ion 520 & Ion 530 ExT Kit-Chef – Thermo Fisher Scientific". thermofisher.com.
- ^ "Raw accuracy". Archived from the original on 30 March 2018. Retrieved 29 March 2018.
- ^ van Vliet AH (1 January 2010). "Next generation sequencing of microbial transcriptomes: challenges and opportunities". FEMS Microbiology Letters. 302 (1): 1–7. doi:10.1111/j.1574-6968.2009.01767.x. PMID 19735299.

- ^ "BGI and MGISEQ". en.mgitech.cn. Retrieved 5 July 2018.
- ^ a b Huang YF, Chen SC, Chiang YS, Chen TH, Chiu KP (2012). "Palindromic sequence impedes sequencing-by-ligation mechanism". BMC Systems Biology. 6 (Suppl 2): S10. doi:10.1186/1752-0509-6-S2-S10. PMC 3521181. PMID 23281822.
- ^ Loose, Matthew; Rakyan, Vardhman; Holmes, Nadine; Payne, Alexander (3 May 2018). "Whale watching with BulkVis: A graphical viewer for Oxford Nanopore bulk fast5 files". bioRxiv 10.1101/312256.
- ^ "PacBio Sales Start to Pick Up as Company Delivers on Product Enhancements". 12 February 2013.
- ^ "Bio-IT World". bio-itworld.com. Archived from the original on 29 July 2020. Retrieved 16 November 2015.
- ^ "PacBio Launches Higher-Throughput, Lower-Cost Single-Molecule Sequencing System". October 2015.
- ^ Clarke J, Wu HC, Jayasinghe L, Patel A, Reid S, Bayley H (April 2009). "Continuous base identification for single-molecule nanopore DNA sequencing". Nature Nanotechnology. 4 (4): 265–70. Bibcode:2009NatNa...4..265C. doi:10.1038/nnano.2009.12. PMID 19350039.
- ^ a b dela Torre R, Larkin J, Singer A, Meller A (2012). "Fabrication and characterization of solid-state nanopore arrays for high-throughput DNA sequencing". Nanotechnology. 23 (38): 385308. Bibcode:2012Nanot..23L5308D. doi:10.1088/0957-4484/23/38/385308. PMC 3557807. PMID 22948520.
- ^ a b Pathak B, Lofas H, Prasongkit J, Grigoriev A, Ahuja R, Scheicher RH (2012). "Double-functionalized nanopore-embedded gold electrodes for rapid DNA sequencing". Applied Physics Letters. 100 (2): 023701. Bibcode:2012ApPhL.100b3701P. doi:10.1063/1.3673335.
- ^ Korlach J, Marks PJ, Cicero RL, Gray JJ, Murphy DL, Roitman DB, Pham TT, Otto GA, Foquet M, Turner SW (2008). "Selective aluminum passivation for targeted immobilization of single DNA polymerase molecules in zero-mode waveguide nanostructures". Proceedings of the National Academy of Sciences. 105 (4): 1176–81. Bibcode:2008PNAS..105.1176K. doi:10.1073/pnas.0710982105. PMC 2234111. PMID 18216253.
- ^ a b Shendure J, Porreca GJ, Reppas NB, Lin X, McCutcheon JP, Rosenbaum AM, Wang MD, Zhang K, Mitra RD, Church GM (9 September 2005). "Accurate multiplex polony sequencing of an evolved bacterial genome". Science. 309 (5741): 1728–32. Bibcode:2005Sci...309.1728S. doi:10.1126/science.1117389. PMID 16081699. S2CID 11405973.
- ^ Bentley DR, Balasubramanian S, et al. (2008). "Accurate whole human genome sequencing using reversible terminator chemistry". Nature. 456 (7218): 53–59. Bibcode:2008Natur.456...53B. doi:10.1038/nature07517. PMC 2581791. PMID 18987734.
- ^ Canard B, Sarfati S (13 October 1994), Novel derivatives usable for the sequencing of nucleic acids, retrieved 9 March 2016
- ^ Canard B, Sarfati RS (October 1994). "DNA polymerase fluorescent substrates with reversible 3'-tags". Gene. 148 (1): 1–6. doi:10.1016/0378-1119(94)90226-7. PMID 7523248.
- ^ Mardis ER (2008). "Next-generation DNA sequencing methods". Annu Rev Genom Hum Genet. 9: 387–402. doi:10.1146/annurev.genom.9.081307.164359. PMID 18576944.
- ^ a b c Drmanac R, Sparks AB, Callow MJ, Halpern AL, Burns NL, Kermani BG, et al. (January 2010). "Human genome sequencing using unchained base reads on self-assembling DNA nanoarrays". Science. 327 (5961): 78–81. Bibcode:2010Sci...327...78D. doi:10.1126/science.1181498. PMID 19892942. S2CID 17309571.
- ^ brandonvd. "About Us – Complete Genomics". Complete Genomics. Retrieved 2 July 2018.
- ^ a b Huang J, Liang X, Xuan Y, Geng C, Li Y, Lu H, et al. (May 2017). "A reference human genome dataset of the BGISEQ-500 sequencer". GigaScience. 6 (5): 1–9. doi:10.1093/gigascience/gix024. PMC 5467036. PMID 28379488.
- ^ Valouev A, Ichikawa J, Tonthat T, Stuart J, Ranade S, Peckham H, Zeng K, Malek JA, Costa G, McKernan K, Sidow A, Fire A, Johnson SM (July 2008). "A high-resolution, nucleosome position map of C. elegans reveals a lack of universal sequence-dictated positioning". Genome Res. 18 (7): 1051–63. doi:10.1101/gr.076463.108. PMC 2493394. PMID 18477713.
- ^ Rusk N (2011). "Torrents of sequence". Nat Methods. 8 (1): 44. doi:10.1038/nmeth.f.330. S2CID 41040192.
- ^ a b Drmanac R, Sparks AB, et al. (2010). "Human Genome Sequencing Using Unchained Base Reads in Self-Assembling DNA Nanoarrays". Science. 327 (5961): 78–81. Bibcode:2010Sci...327...78D. doi:10.1126/science.1181498. PMID 19892942. S2CID 17309571.
- ^ Porreca GJ (2010). "Genome Sequencing on Nanoballs". Nature Biotechnology. 28 (1): 43–44. doi:10.1038/nbt0110-43. PMID 20062041. S2CID 54557996.
- ^ "HeliScope Gene Sequencing / Genetic Analyzer System : Helicos BioSciences". 2 November 2009. Archived from the original on 2 November 2009.
- ^ Thompson JF, Steinmann KE (October 2010). "Single molecule sequencing with a HeliScope genetic analysis system". Current Protocols in Molecular Biology. Chapter 7: Unit7.10. doi:10.1002/0471142727.mb0710s92. PMC 2954431. PMID 20890904.
- ^ "tSMS SeqLL Technical Explanation". SeqLL. Archived from the original on 8 August 2014. Retrieved 9 August 2015.
- ^ Heather, James M.; Chain, Benjamin (January 2016). "The sequence of sequencers: The history of sequencing DNA". Genomics. 107 (1): 1–8. doi:10.1016/j.ygeno.2015.11.003. PMC 4727787. PMID 26554401.
- ^ Sara El-Metwally; Osama M. Ouda; Mohamed Helmy (2014). "New Horizons in Next-Generation Sequencing". Next Generation Sequencing Technologies and Challenges in Sequence Assembly. SpringerBriefs in Systems Biology. Vol. 7. Next Generation Sequencing Technologies and Challenges in Sequence Assembly, Springer Briefs in Systems Biology Volume 7. pp. 51–59. doi:10.1007/978-1-4939-0715-1_6. ISBN 978-1-4939-0714-4.
- ^ a b Fair RB, Khlystov A, Tailor TD, Ivanov V, Evans RD, Srinivasan V, Pamula VK, Pollack MG, Griffin PB, Zhou J (January 2007). "Chemical and Biological Applications of Digital-Microfluidic Devices". IEEE Design & Test of Computers. 24 (1): 10–24. CiteSeerX 10.1.1.559.1440. doi:10.1109/MDT.2007.8. hdl:10161/6987. S2CID 10122940.
- ^ a b Boles DJ, Benton JL, Siew GJ, Levy MH, Thwar PK, Sandahl MA, et al. (November 2011). "Droplet-based pyrosequencing using digital microfluidics". Analytical Chemistry. 83 (22): 8439–47. doi:10.1021/ac201416j. PMC 3690483. PMID 21932784.
- ^ Zilionis R, Nainys J, Veres A, Savova V, Zemmour D, Klein AM, Mazutis L (January 2017). "Single-cell barcoding and sequencing using droplet microfluidics". Nature Protocols. 12 (1): 44–73. doi:10.1038/nprot.2016.154. PMID 27929523. S2CID 767782.
- ^ "The Harvard Nanopore Group". Mcb.harvard.edu. Archived from the original on 21 February 2002. Retrieved 15 November 2009.
- ^ "Nanopore Sequencing Could Slash DNA Analysis Costs".
- ^ 미국 특허 20060029957, ZS Genetics, "핵산 고분자 및 관련 성분 분석 시스템 및 방법", 2005-07-14호 발행
- ^ Xu M, Fujita D, Hanagata N (December 2009). "Perspectives and challenges of emerging single-molecule DNA sequencing technologies". Small. 5 (23): 2638–49. doi:10.1002/smll.200900976. PMID 19904762.
- ^ Schadt EE, Turner S, Kasarskis A (2010). "A window into third-generation sequencing". Human Molecular Genetics. 19 (R2): R227–40. doi:10.1093/hmg/ddq416. PMID 20858600.
- ^ Xu M, Endres RG, Arakawa Y (2007). "The electronic properties of DNA bases". Small. 3 (9): 1539–43. doi:10.1002/smll.200600732. PMID 17786897.
- ^ Di Ventra M (2013). "Fast DNA sequencing by electrical means inches closer". Nanotechnology. 24 (34): 342501. Bibcode:2013Nanot..24H2501D. doi:10.1088/0957-4484/24/34/342501. PMID 23899780. S2CID 140101884.
- ^ Ohshiro T, Matsubara K, Tsutsui M, Furuhashi M, Taniguchi M, Kawai T (2012). "Single-molecule electrical random resequencing of DNA and RNA". Sci Rep. 2: 501. Bibcode:2012NatSR...2E.501O. doi:10.1038/srep00501. PMC 3392642. PMID 22787559.
- ^ Hanna GJ, Johnson VA, Kuritzkes DR, Richman DD, Martinez-Picado J, Sutton L, Hazelwood JD, D'Aquila RT (1 July 2000). "Comparison of Sequencing by Hybridization and Cycle Sequencing for Genotyping of Human Immunodeficiency Virus Type 1 Reverse Transcriptase". J. Clin. Microbiol. 38 (7): 2715–21. doi:10.1128/JCM.38.7.2715-2721.2000. PMC 87006. PMID 10878069.
- ^ a b Morey M, Fernández-Marmiesse A, Castiñeiras D, Fraga JM, Couce ML, Cocho JA (2013). "A glimpse into past, present, and future DNA sequencing". Molecular Genetics and Metabolism. 110 (1–2): 3–24. doi:10.1016/j.ymgme.2013.04.024. PMID 23742747.
- ^ Qin Y, Schneider TM, Brenner MP (2012). Gibas C (ed.). "Sequencing by Hybridization of Long Targets". PLOS ONE. 7 (5): e35819. Bibcode:2012PLoSO...735819Q. doi:10.1371/journal.pone.0035819. PMC 3344849. PMID 22574124.
- ^ Edwards JR, Ruparel H, Ju J (2005). "Mass-spectrometry DNA sequencing". Mutation Research. 573 (1–2): 3–12. doi:10.1016/j.mrfmmm.2004.07.021. PMID 15829234.
- ^ Hall TA, Budowle B, Jiang Y, Blyn L, Eshoo M, Sannes-Lowery KA, Sampath R, Drader JJ, Hannis JC, Harrell P, Samant V, White N, Ecker DJ, Hofstadler SA (2005). "Base composition analysis of human mitochondrial DNA using electrospray ionization mass spectrometry: A novel tool for the identification and differentiation of humans". Analytical Biochemistry. 344 (1): 53–69. doi:10.1016/j.ab.2005.05.028. PMID 16054106.
- ^ Howard R, Encheva V, Thomson J, Bache K, Chan YT, Cowen S, Debenham P, Dixon A, Krause JU, Krishan E, Moore D, Moore V, Ojo M, Rodrigues S, Stokes P, Walker J, Zimmermann W, Barallon R (15 June 2011). "Comparative analysis of human mitochondrial DNA from World War I bone samples by DNA sequencing and ESI-TOF mass spectrometry". Forensic Science International: Genetics. 7 (1): 1–9. doi:10.1016/j.fsigen.2011.05.009. PMID 21683667.
- ^ Monforte JA, Becker CH (1 March 1997). "High-throughput DNA analysis by time-of-flight mass spectrometry". Nature Medicine. 3 (3): 360–62. doi:10.1038/nm0397-360. PMID 9055869. S2CID 28386145.
- ^ Beres SB, Carroll RK, Shea PR, Sitkiewicz I, Martinez-Gutierrez JC, Low DE, McGeer A, Willey BM, Green K, Tyrrell GJ, Goldman TD, Feldgarden M, Birren BW, Fofanov Y, Boos J, Wheaton WD, Honisch C, Musser JM (8 February 2010). "Molecular complexity of successive bacterial epidemics deconvoluted by comparative pathogenomics". Proceedings of the National Academy of Sciences. 107 (9): 4371–76. Bibcode:2010PNAS..107.4371B. doi:10.1073/pnas.0911295107. PMC 2840111. PMID 20142485.
- ^ Kan CW, Fredlake CP, Doherty EA, Barron AE (1 November 2004). "DNA sequencing and genotyping in miniaturized electrophoresis systems". Electrophoresis. 25 (21–22): 3564–88. doi:10.1002/elps.200406161. PMID 15565709. S2CID 4851728.
- ^ Chen YJ, Roller EE, Huang X (2010). "DNA sequencing by denaturation: experimental proof of concept with an integrated fluidic device". Lab on a Chip. 10 (9): 1153–59. doi:10.1039/b921417h. PMC 2881221. PMID 20390134.
- ^ Bell DC, Thomas WK, Murtagh KM, Dionne CA, Graham AC, Anderson JE, Glover WR (9 October 2012). "DNA Base Identification by Electron Microscopy". Microscopy and Microanalysis. 18 (5): 1049–53. Bibcode:2012MiMic..18.1049B. doi:10.1017/S1431927612012615. PMID 23046798. S2CID 25713635.
- ^ Pareek CS, Smoczynski R, Tretyn A (November 2011). "Sequencing technologies and genome sequencing". Journal of Applied Genetics. 52 (4): 413–35. doi:10.1007/s13353-011-0057-x. PMC 3189340. PMID 21698376.
- ^ Pareek CS, Smoczynski R, Tretyn A (2011). "Sequencing technologies and genome sequencing". Journal of Applied Genetics. 52 (4): 413–35. doi:10.1007/s13353-011-0057-x. PMC 3189340. PMID 21698376.
- ^ Fujimori S, Hirai N, Ohashi H, Masuoka K, Nishikimi A, Fukui Y, Washio T, Oshikubo T, Yamashita T, Miyamoto-Sato E (2012). "Next-generation sequencing coupled with a cell-free display technology for high-throughput production of reliable interactome data". Scientific Reports. 2: 691. Bibcode:2012NatSR...2E.691F. doi:10.1038/srep00691. PMC 3466446. PMID 23056904.
- ^ "2022 Sequencing Market Share – Same as It Ever Was (For Now)". 25 June 2023.
- ^ Harbers M (2008). "The Current Status of cDNA Cloning". Genomics. 91 (3): 232–42. doi:10.1016/j.ygeno.2007.11.004. PMID 18222633.
- ^ Alberti A, Belser C, Engelen S, Bertrand L, Orvain C, Brinas L, Cruaud C, et al. (2014). "Comparison of Library Preparation Methods Reveals Their Impact on Interpretation of Metatranscriptomic Data". BMC Genomics. 15 (1): 912–12. doi:10.1186/1471-2164-15-912. PMC 4213505. PMID 25331572.
- ^ "Scalable Nucleic Acid Quality Assessments for Illumina Next-Generation Sequencing Library Prep" (PDF). Retrieved 27 December 2017.
- ^ "Archon Genomics XPRIZE". Archon Genomics XPRIZE. Archived from the original on 17 June 2013. Retrieved 9 August 2007.
- ^ "Grant Information". National Human Genome Research Institute (NHGRI).
- ^ Severin J, Lizio M, Harshbarger J, Kawaji H, Daub CO, Hayashizaki Y, Bertin N, Forrest AR (2014). "Interactive visualization and analysis of large-scale sequencing datasets using ZENBU". Nat. Biotechnol. 32 (3): 217–19. doi:10.1038/nbt.2840. PMID 24727769. S2CID 26575621.
- ^ Shmilovici A, Ben-Gal I (2007). "Using a VOM model for reconstructing potential coding regions in EST sequences" (PDF). Computational Statistics. 22 (1): 49–69. doi:10.1007/s00180-007-0021-8. S2CID 2737235. Archived from the original (PDF) on 31 May 2020. Retrieved 10 January 2014.
- ^ Del Fabbro C, Scalabrin S, Morgante M, Giorgi FM (2013). "An Extensive Evaluation of Read Trimming Effects on Illumina NGS Data Analysis". PLOS ONE. 8 (12): e85024. Bibcode:2013PLoSO...885024D. doi:10.1371/journal.pone.0085024. PMC 3871669. PMID 24376861.
- ^ Martin, Marcel (2 May 2011). "Cutadapt removes adapter sequences from high-throughput sequencing reads". EMBnet.journal. 17 (1): 10. doi:10.14806/ej.17.1.200.
- ^ Smeds L, Künstner A (19 October 2011). "ConDeTri--a content dependent read trimmer for Illumina data". PLOS ONE. 6 (10): e26314. Bibcode:2011PLoSO...626314S. doi:10.1371/journal.pone.0026314. PMC 3198461. PMID 22039460.
- ^ Prezza N, Del Fabbro C, Vezzi F, De Paoli E, Policriti A (2012). "Erne-Bs5". Proceedings of the ACM Conference on Bioinformatics, Computational Biology and Biomedicine. Vol. 12. pp. 12–19. doi:10.1145/2382936.2382938. ISBN 9781450316705. S2CID 5673753.
- ^ Schmieder R, Edwards R (March 2011). "Quality control and preprocessing of metagenomic datasets". Bioinformatics. 27 (6): 863–4. doi:10.1093/bioinformatics/btr026. PMC 3051327. PMID 21278185.
- ^ Bolger AM, Lohse M, Usadel B (August 2014). "Trimmomatic: a flexible trimmer for Illumina sequence data". Bioinformatics. 30 (15): 2114–20. doi:10.1093/bioinformatics/btu170. PMC 4103590. PMID 24695404.
- ^ Cox MP, Peterson DA, Biggs PJ (September 2010). "SolexaQA: At-a-glance quality assessment of Illumina second-generation sequencing data". BMC Bioinformatics. 11 (1): 485. doi:10.1186/1471-2105-11-485. PMC 2956736. PMID 20875133.
- ^ Murray TH (January 1991). "Ethical issues in human genome research". FASEB Journal. 5 (1): 55–60. doi:10.1096/fasebj.5.1.1825074. PMID 1825074. S2CID 20009748.
- ^ a b c Robertson JA (August 2003). "The $1000 genome: ethical and legal issues in whole genome sequencing of individuals". The American Journal of Bioethics. 3 (3): W–IF1. doi:10.1162/152651603322874762. PMID 14735880. S2CID 15357657.
- ^ a b Henderson, Mark (9 September 2013). "Human genome sequencing: the real ethical dilemmas". The Guardian. Retrieved 20 May 2015.
- ^ Harmon, Amy (24 February 2008). "Insurance Fears Lead Many to Shun DNA Tests". The New York Times. Retrieved 20 May 2015.
- ^ 2007년 4월 27일 관리예산국 대통령실 행정정책명세서
- ^ National Human Genome Research Institute (21 May 2008). "President Bush Signs the Genetic Information Nondiscrimination Act of 2008". Retrieved 17 February 2014.
- ^ Baker, Monya (11 October 2012). "US ethics panel reports on DNA sequencing and privacy". Nature News Blog.
- ^ "Privacy and Progress in Whole Genome Sequencing" (PDF). Presidential Commission for the Study of Bioethical Issues. Archived from the original (PDF) on 12 June 2015. Retrieved 20 May 2015.
- ^ Hartnett, Kevin (12 May 2013). "The DNA in your garbage: up for grabs". The Boston Globe. Retrieved 2 January 2023.
- ^ Goldenberg AJ, Sharp RR (February 2012). "The ethical hazards and programmatic challenges of genomic newborn screening". JAMA. 307 (5): 461–2. doi:10.1001/jama.2012.68. PMC 3868436. PMID 22298675.
- ^ Hughes, Virginia (7 January 2013). "It's Time To Stop Obsessing About the Dangers of Genetic Information". Slate Magazine. Retrieved 22 May 2015.
- ^ a b Bloss CS, Schork NJ, Topol EJ (February 2011). "Effect of direct-to-consumer genomewide profiling to assess disease risk". The New England Journal of Medicine. 364 (6): 524–34. doi:10.1056/NEJMoa1011893. PMC 3786730. PMID 21226570.
- ^ Rochman, Bonnie (25 October 2012). "What Your Doctor Isn't Telling You About Your DNA". Time.com. Retrieved 22 May 2015.
- ^ Sajeer P, Muhammad (4 May 2023). "Disruptive technology: Exploring the ethical, legal, political, and societal implications of nanopore sequencing technology". EMBO Reports. 24 (5): e56619. doi:10.15252/embr.202256619. PMC 10157308. PMID 36988424. S2CID 257803254.
외부 링크
| 라이브러리 리소스 정보 DNA 염기서열 분석 |


