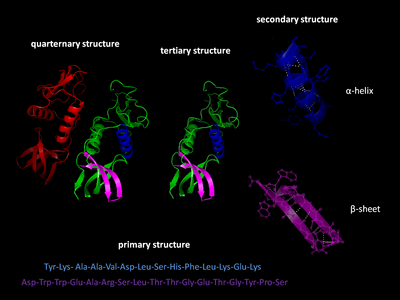
단백질 구조
Protein structure단백질 구조는 아미노산 사슬 분자의 원자의 3차원 배열이다.단백질은 고분자, 특히 폴리펩타이드로, 고분자의 모노머인 아미노산의 배열로 형성됩니다.단일 아미노산 단량체는 폴리머의 반복 단위를 나타내는 잔기라고도 할 수 있다.단백질은 축합 반응을 하는 아미노산에 의해 형성되는데, 아미노산은 펩타이드 결합으로 서로 결합하기 위해 반응당 하나의 물 분자를 잃는다.관례상 30개 이하의 아미노산 사슬은 [1]단백질이 아닌 펩타이드로 식별되는 경우가 많다.그들의 생물학적 기능을 수행하기 위해, 단백질은 수소 결합, 이온 상호작용, 반데르발스 힘, 그리고 소수성 패킹과 같은 많은 비공유 상호작용에 의해 움직이는 하나 이상의 특정한 공간적 배치로 접힙니다.분자 수준에서 단백질의 기능을 이해하기 위해서는 종종 단백질의 3차원 구조를 결정할 필요가 있다.이것은 단백질의 구조를 결정하기 위해 X선 결정학, NMR 분광학, 크라이오 전자 현미경법(cryo-EM), 이중 편파 간섭법과 같은 기술을 사용하는 구조 생물학 과학 분야의 주제이다.
단백질 구조는 크기가 수십에서 수천 개의 아미노산까지 [2]다양합니다.물리적 크기에 따라 단백질은 1~100nm의 나노입자로 분류된다.단백질 소단위로부터 매우 큰 단백질 복합체가 형성될 수 있다.예를 들어, 수천 개의 액틴 분자가 미세 필라멘트로 조립됩니다.
단백질은 보통 생물학적 기능을 수행함에 있어 가역적인 구조적 변화를 겪는다.같은 단백질의 대체 구조는 다른 형태라고 불리며, 그들 사이의 전이는 형태 변화라고 불립니다.
단백질 구조 수준
단백질 구조에는 4가지 뚜렷한 레벨이 있습니다.
일차 구조
단백질의 1차 구조는 폴리펩타이드 사슬에 있는 아미노산의 염기서열을 말한다.1차 구조는 단백질 생합성 과정에서 만들어지는 펩타이드 결합에 의해 결합된다.폴리펩타이드 사슬의 양 끝은 각 끝의 활성기의 성질에 따라 카르복실 말단(C-terminus)과 아미노 말단(N-terminus)으로 지칭된다.잔류물의 계수는 항상 아미노기가 펩타이드 결합에 관여하지 않는 N-말단(NH-group2)에서 시작된다.단백질의 1차 구조는 단백질에 대응하는 유전자에 의해 결정된다.DNA에 있는 뉴클레오티드의 특정 배열은 mRNA로 전사되고, 리보솜은 번역이라고 불리는 과정에서 리보솜에 의해 읽힌다.인슐린의 아미노산 배열은 프레데릭 생어에 의해 발견되었고, 단백질은 아미노산 [3][4]배열을 정의한다는 것을 증명했다.단백질의 배열은 해당 단백질에 고유하며 단백질의 구조와 기능을 정의합니다.단백질의 배열은 에드만 분해 또는 탠덤 질량 분석과 같은 방법으로 결정될 수 있다.그러나 종종 유전자 코드를 사용하여 유전자의 배열에서 직접 읽힌다.펩타이드 결합이 형성되면 물 분자가 손실되어 단백질이 아미노산 잔류물로 구성되기 때문에 단백질을 논할 때 아미노산 잔류물이라는 단어를 사용하는 것이 엄격히 권장된다.인산화 및 글리코실화와 같은 번역 후 변형도 보통 1차 구조의 일부로 간주되며 유전자에서 읽을 수 없다.예를 들어 인슐린은 2개의 사슬에 51개의 아미노산으로 구성되어 있습니다.한 사슬은 31개의 아미노산을 가지고 있고 다른 사슬은 20개의 아미노산을 가지고 있다.
이차 구조
2차 구조는 실제 폴리펩타이드 백본 체인상의 매우 규칙적인 국소 서브 구조를 말한다.2차 구조의 두 가지 주요 유형인 [5]α-나선과 β-스트랜드 또는 β-시트는 1951년 라이너스 폴링 외 연구진에 의해 제안되었다.이러한 2차 구조는 주쇄 펩타이드 그룹 사이의 수소 결합 패턴에 의해 정의된다.이들은 라마찬드란 그림에서 이면각 θ와 θ의 특정 값으로 구속되는 규칙적인 형상을 가지고 있다.α-나선 및 β-시트 모두 펩타이드 골격 내의 모든 수소 결합 공여체 및 수용체를 포화시키는 방법을 나타낸다.단백질의 일부는 순서가 매겨져 있지만 규칙적인 구조를 형성하지는 않는다.이들은 고정된 3차원 구조가 없는 전개된 폴리펩타이드 체인인 랜덤 코일과 혼동해서는 안 됩니다.여러 개의 순차적 이차 구조가 "초 이차 단위"[6]를 형성할 수 있다.
3차 구조
3차 구조는 단일 단백질 분자(단일 폴리펩타이드 사슬)에 의해 생성된 3차원 구조를 말한다.하나 또는 여러 개의 도메인을 포함할 수 있습니다.α-헬릭스와 β-플리티드 시트는 콤팩트한 구상 구조로 접혀 있다.접힘은 비특이적 소수성 상호작용, 물에서 나오는 소수성 잔류물의 매립에 의해 추진되지만, 염교, 수소 결합, 측쇄와 디황화물 결합의 밀착과 같은 특정 3차 상호작용에 의해 단백질 도메인의 일부가 제자리에 고정될 때만 구조가 안정적이다.디술피드 결합은 세포 내 액체(cytosol)가 일반적으로 환원 환경이기 때문에 세포질 단백질에서 매우 드물다.
4차 구조
4차 구조는 단일 기능 단위(멀티머)로서 동작하는 2개 이상의 개별 폴리펩타이드 사슬(서브유닛)의 집합으로 이루어진 3차원 구조이다.생성된 멀티머는 3차 구조와 동일한 비공유 상호작용 및 디술피드 결합에 의해 안정화된다.많은 가능한 4차 구조 [7]조직들이 있다.2개 이상의 폴리펩타이드 복합체(즉, 복수의 서브유닛)를 멀티머라고 한다.구체적으로는 2개의 서브유닛, 3개의 서브유닛, 4개의 서브유닛, 5개의 서브유닛을 포함하는 4개의 서브유닛, 5개의 서브유닛을 포함하는 5개의 펜타머를 포함하는 것을 다이머라고 합니다.서브유닛은 다이머의 2중축과 같은 대칭 연산에 의해 서로 자주 관련지어집니다.동일한 서브유닛으로 구성된 멀티머는 접두사 "homo-"로, 다른 서브유닛으로 구성된 멀티머는 접두사 "hetero-"로 참조된다. 예를 들어 헤모글로빈의 2개의 알파 및 2개의 베타사슬과 같은 헤테로테로-"로 참조된다.
단백질 구조의 영역, 모티브 및 주름
단백질은 종종 몇 가지 구조 단위로 구성되어 있다고 묘사된다.이러한 단위에는 도메인, 모티브 및 접힘이 포함됩니다.진핵생물계에서 발현되는 약 100,000개의 다른 단백질이 있다는 사실에도 불구하고, 훨씬 적은 수의 다른 영역, 구조적 모티브 그리고 접힘이 있다.
구조 영역
구조 도메인은 단백질의 전체 구조의 요소이며, 자기 안정적이며 종종 단백질 사슬의 나머지 부분과 독립적으로 접힌다.많은 도메인이 한 유전자 또는 한 유전자 패밀리의 단백질 생성물에 고유한 것이 아니라 다양한 단백질에 나타납니다.도메인은 종종 그들이 속한 단백질의 생물학적 기능(예: 칼모듈린의 칼슘 결합 도메인)에서 중요한 역할을 하기 때문에 명명되고 선택된다.그것들은 독립적으로 안정적이기 때문에, 도메인은 키메라 단백질을 만들기 위해 한 단백질과 다른 단백질 사이의 유전 공학에 의해 "교환"될 수 있다.단백질 티로신 포스파타아제 도메인과 C2 도메인 쌍과 같이 서로 다른 단백질에서 발생하는 여러 도메인의 보수적인 조합을 단일 [8]단위로 진화할 수 있는 "슈퍼 도메인"이라고 불렀다.
구조 및 시퀀스 모티브
구조 및 배열 모티브는 많은 다른 단백질에서 발견된 단백질 3차원 구조 또는 아미노산 배열의 짧은 세그먼트를 참조한다.
초이차 구조
3차 단백질 구조는 동일한 폴리펩타이드 사슬에 여러 2차 요소를 가질 수 있다.2차 구조는 β-α-β 단위 또는 나선-턴-나선 모티브와 같은 2차 구조 요소의 특정 조합을 의미한다.그 중 일부는 구조적 모티브라고도 할 수 있다.
단백질 접힘
단백질 접힘은 단백질 구조 [9]분류 데이터베이스에 제공된 나선 다발, β-배럴, 로스만 접힘 또는 다른 "접힘"과 같은 일반적인 단백질 구조를 말한다.관련된 개념은 단백질 위상이다.
단백질 역학 및 입체구조 앙상블
단백질은 정적인 물체가 아니라 입체구조 상태의 앙상블을 채운다.이러한 상태 사이의 전환은 일반적으로 나노스케일에서 발생하며, 알로스테릭[10] 신호 전달 및 효소 [11]촉매 작용과 같은 기능적으로 관련된 현상과 관련이 있다.단백질 역학과 구조 변화는 단백질이 종종 다단백질 [12]복합체의 형태로 세포 내에서 나노 크기의 생물학적 기계로 기능하도록 합니다.예를 들어 근육 수축을 담당하는 미오신, 세포 내부의 화물을 미세관을 따라 세포에서 핵으로 이동시키는 키네신, 세포 내부의 화물을 핵으로 이동시키고 운동섬모와 편모의 축삭 박동을 생성하는 다이네인과 같은 운동단백질이 포함된다.[I]n 효과, [모바일 섬모]는 분자 복합체에 600개 이상의 단백질로 구성된 나노 기계이며, 이들 중 다수는 나노 기계로도 독립적으로 기능합니다.유연한 링커는 그들에 의해 연결된 이동 단백질 도메인이 그들의 결합 파트너를 모집하고 단백질 도메인 역학을 통해 장거리 알로스테리를 유도할 수 있게 한다."[13]
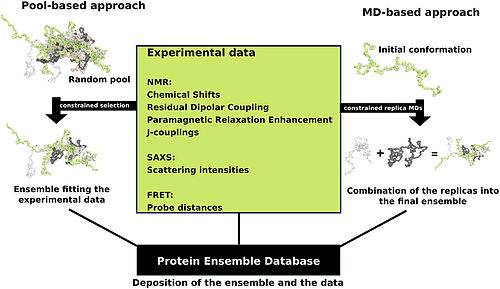
단백질은 종종 다른 단백질과의 상호작용에 의해 영향을 받은 후 입체구조 변화를 경험하는 비교적 안정적인 3차 구조 또는 효소 활동의 일부로 생각됩니다.그러나 단백질은 안정성이 다를 수 있으며, 안정성이 떨어지는 변종 중 일부는 본질적으로 무질서한 단백질이다.이들 단백질은 안정적인 3차 구조가 없는 비교적 '무질서' 상태에서 존재하며 기능한다.그 결과 단일 고정 3차 구조로 기술하는 것은 어렵다.입체구조 앙상블은 본질적으로 무질서한 [15][14]단백질의 입체구조 상태를 보다 정확하고 '동적인' 표현으로 제공하기 위한 방법으로 고안되었다.
단백질 앙상블 파일은 유연한 구조를 가진 것으로 간주될 수 있는 단백질의 표현이다.이러한 파일을 작성하려면 이론적으로 가능한 다양한 단백질 구성 중 실제로 존재하는 것을 결정해야 합니다.한 가지 접근방식은 단백질 데이터에 연산 알고리즘을 적용하여 앙상블 파일에 가장 가능성이 높은 일련의 구성을 결정하는 것입니다.단백질 앙상블 데이터베이스에는 풀과 분자역학(MD) 접근방식의 두 가지 일반적인 방법론(그림에 나타낸 그림)으로 분류되는 여러 가지 방법이 있다.풀 기반 접근법은 단백질의 아미노산 배열을 사용하여 대량의 무작위 배열을 만듭니다.그런 다음 이 풀은 구조에 따라 각 구성에 대한 이론적 매개 변수 세트를 생성하는 더 많은 계산 처리를 거칩니다.평균 이론 매개변수가 이 단백질에 대해 알려진 실험 데이터와 밀접하게 일치하는 이 풀의 입체구조 서브셋이 선택된다.대안 분자 역학 접근법은 한 번에 여러 개의 무작위 배열을 취하며 모든 배열을 실험 데이터에 적용시킨다.여기서 실험 데이터는 구성(예를 들어 원자 사이의 알려진 거리)에 배치되는 한계로 작용한다.실험 데이터에 의해 설정된 한계 내에서 유지되는 구성만 허용됩니다.이 접근방식은 종종 많은 양의 실험 데이터를 매우 계산적으로 까다로운 [14]작업에 적용한다.
입체구조 앙상블은 Sic1/Cdc4,[16] p15 PAF,[17] MKK7,[18] 베타-시뉴클레인[19] 및 P27과[20] 같은 고도로 역동적이고 부분적으로 전개된 단백질에 대해 생성되었다.
단백질 폴딩
이 섹션은 확장해야 합니다.추가해서 도와주실 수 있습니다. (2019년 4월) |
번역될 때 폴리펩타이드는 대부분 무작위 코일로 리보솜을 빠져나와 원래 상태로 [21][22]접힙니다.단백질 사슬의 최종 구조는 일반적으로 아미노산 배열(Anfinsen의 도그마)[23]에 의해 결정된다고 가정한다.
단백질 안정성
단백질의 열역학적 안정성은 접힌 단백질 상태와 펴진 단백질 상태 사이의 자유 에너지 차이를 나타냅니다.이 자유 에너지 차이는 온도에 매우 민감하기 때문에 온도의 변화로 인해 전개 또는 변성이 발생할 수 있습니다.단백질 변성은 기능 상실과 자연 상태의 상실을 초래할 수 있다.용해성 구상 단백질의 안정화 자유 에너지는 일반적으로 50 kJ/[citation needed]mol을 초과하지 않는다.2차 구조의 안정화와 소수성 상호작용에 의한 내부 코어 안정화를 위해 일어나는 수소 결합의 수가 많은 것을 고려하면 안정화의 자유 에너지는 큰 [24]수와의 작은 차이로 나타난다.
단백질 구조 결정

단백질 데이터 뱅크에서 사용할 수 있는 단백질 구조의 약 90%가 X선 결정학에 [25]의해 결정되었습니다.결정화된 단백질 속 전자의 3차원(3D) 밀도 분포를 측정해 특정 분해능으로 모든 원자의 3차원 좌표를 유추할 수 있다.알려진 단백질 구조의 약 9%는 핵자기공명([citation needed]NMR) 기술에 의해 얻어졌다.더 큰 단백질 복합체의 경우, 저온 전자 현미경 검사를 통해 단백질 구조를 결정할 수 있습니다.분해능은 일반적으로 X선 결정학(NMR)보다 낮지만 최대 분해능은 꾸준히 증가하고 있습니다.이 기술은 바이러스 피복 단백질과 아밀로이드 섬유와 같은 매우 큰 단백질 복합체들에게 여전히 특히 중요하다.
일반적인 2차 구조 조성물은 원형 이색성을 통해 결정될 수 있다.진동 분광법은 또한 펩타이드, 폴리펩타이드 및 [26]단백질의 구조를 특징짓기 위해 사용될 수 있다.2차원 적외선 분광법은 다른 [27][28]방법으로는 연구할 수 없는 유연한 펩타이드와 단백질의 구조를 조사하는 귀중한 방법이 되었다.단백질 구조의 보다 질적인 그림은 종종 단백질 분해에 의해 얻어지는데, 이것은 또한 보다 결정성이 높은 단백질 샘플을 선별하는 데 유용하다.고속 병렬 단백질 분해(FASTPP)를 포함한 이 접근방식의 새로운 구현은 정제 필요 없이 구조화된 분율과 [29]그 안정성을 조사할 수 있다.일단 단백질의 구조가 실험적으로 결정되면, 그 [30]구조의 분자 역학 시뮬레이션을 사용하여 더 상세한 연구가 계산적으로 이루어질 수 있다.
단백질 구조 데이터베이스
단백질 구조 데이터베이스는 실험적으로 결정된 다양한 단백질 구조를 중심으로 모델화된 데이터베이스이다.대부분의 단백질 구조 데이터베이스의 목적은 단백질 구조를 구성하고 주석을 달아 생물학적 공동체가 유용한 방법으로 실험 데이터에 접근할 수 있도록 하는 것이다.단백질 구조 데이터베이스에 포함된 데이터는 종종 3D 좌표뿐만 아니라 단위 세포 치수 및 X선 결정학적 결정 구조를 위한 각도 등의 실험 정보를 포함한다.대부분의 경우, 단백질 또는 단백질의 특정 구조 결정 또한 시퀀스 정보를 포함하고 일부 데이터베이스는 시퀀스 기반 쿼리를 수행하기 위한 수단을 제공하지만, 구조 데이터베이스의 주요 속성은 구조 정보인 반면 시퀀스 데이터베이스는 시퀀스 정보에 초점을 맞춘다.d 대부분의 엔트리에 대한 구조 정보가 포함되어 있지 않다.단백질 구조 데이터베이스는 구조 기반 의약품 설계와 같은 컴퓨터 생물학에서 사용된 계산 방법을 개발하고 [31]단백질의 기능에 대한 통찰력을 제공하기 위해 일부 방법에 의해 사용되는 대규모 실험 데이터 세트를 제공하는 데 있어 매우 중요하다.
단백질의 구조적 분류
단백질 구조는 구조 유사성, 위상 클래스 또는 공통 진화 기원에 따라 분류될 수 있다.단백질[32] 구조 분류 데이터베이스와 CATH[33] 데이터베이스는 단백질의 두 가지 다른 구조 분류를 제공한다.구조적 유사성이 클 때, 두 단백질은 공통의 [34]조상으로부터 분리되었을 가능성이 있으며, 단백질 사이의 공유 구조는 호몰로지의 증거로 간주된다.구조 유사성은 단백질을 단백질 [35]슈퍼패밀리로 그룹화하는 데 사용될 수 있다.공유 구조가 유의하지만 공유 비율이 작을 경우, 공유 단편은 수평 유전자 이동과 같은 보다 극적인 진화 사건의 결과일 수 있으며, 이러한 단편들을 단백질 슈퍼패밀리에 결합하는 것은 더 이상 [34]정당화되지 않는다.단백질의 위상은 단백질을 분류하는데도 사용될 수 있다.매듭 이론과 회로 위상은 각각 체인 교차와 내부 접점에 기초한 단백질 접힘의 분류를 위해 개발된 두 가지 위상 프레임워크입니다.
단백질 구조의 계산 예측
단백질 배열의 생성은 단백질 구조의 결정보다 훨씬 쉽다.그러나 단백질의 구조는 배열보다 단백질의 기능에 대해 훨씬 더 많은 통찰력을 준다.따라서 단백질 구조의 염기서열에서 계산 예측을 위한 많은 방법이 [36]개발되었다.ab initio 예측 방법은 단백질의 배열만을 사용한다.스레드화 및 호몰로지 모델링 방법은 단백질 패밀리라고 불리는 진화와 관련된 단백질의 실험 구조로부터 알려지지 않은 구조의 단백질에 대한 3-D 모델을 구축할 수 있습니다.
「 」를 참조해 주세요.
레퍼런스
- ^ H. Stephen Stoker (1 January 2015). Organic and Biological Chemistry. Cengage Learning. p. 371. ISBN 978-1-305-68645-8.
- ^ Brocchieri L, Karlin S (10 June 2005). "Protein length in eukaryotic and prokaryotic proteomes". Nucleic Acids Research. 33 (10): 3390–3400. doi:10.1093/nar/gki615. PMC 1150220. PMID 15951512.
- ^ Sanger, F.; Tuppy, H. (1 September 1951). "The amino-acid sequence in the phenylalanyl chain of insulin. I. The identification of lower peptides from partial hydrolysates". The Biochemical Journal. 49 (4): 463–481. doi:10.1042/bj0490463. ISSN 0264-6021. PMC 1197535. PMID 14886310.
- ^ Sanger, F. (15 May 1959). "Chemistry of Insulin". Science. 129 (3359): 1340–1344. Bibcode:1959Sci...129.1340G. doi:10.1126/science.129.3359.1340. ISSN 0036-8075. PMID 13658959.
- ^ Pauling L, Corey RB, Branson HR (1951). "The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain". Proc Natl Acad Sci USA. 37 (4): 205–211. Bibcode:1951PNAS...37..205P. doi:10.1073/pnas.37.4.205. PMC 1063337. PMID 14816373.
- ^ Chiang YS, Gelfand TI, Kister AE, Gelfand IM (2007). "New classification of supersecondary structures of sandwich-like proteins uncovers strict patterns of strand assemblage". Proteins. 68 (4): 915–921. doi:10.1002/prot.21473. PMID 17557333. S2CID 29904865.
- ^ Moutevelis E, Woolfson DN (January 2009). "A periodic table of coiled-coil protein structures". J. Mol. Biol. 385 (3): 726–32. doi:10.1016/j.jmb.2008.11.028. ISSN 0022-2836. PMID 19059267.
- ^ Haynie DT, Xue B (2015). "Superdomain in the protein structure hierarchy: the case of PTP-C2". Protein Science. 24 (5): 874–82. doi:10.1002/pro.2664. PMC 4420535. PMID 25694109.
- ^ Govindarajan S, Recabarren R, Goldstein RA (17 September 1999). "Estimating the total number of protein folds". Proteins. 35 (4): 408–414. doi:10.1002/(SICI)1097-0134(19990601)35:4<408::AID-PROT4>3.0.CO;2-A. hdl:2027.42/34969. PMID 10382668. Archived from the original on 5 January 2013.
- ^ Bu Z, Callaway DJ (2011). "Proteins MOVE! Protein dynamics and long-range allostery in cell signaling". Protein Structure and Diseases. Advances in Protein Chemistry and Structural Biology. Vol. 83. Academic Press. pp. 163–221. doi:10.1016/B978-0-12-381262-9.00005-7. ISBN 9780123812629. PMID 21570668.
- ^ Fraser JS, Clarkson MW, Degnan SC, Erion R, Kern D, Alber T (December 2009). "Hidden alternative structures of proline isomerase essential for catalysis". Nature. 462 (7273): 669–673. Bibcode:2009Natur.462..669F. doi:10.1038/nature08615. PMC 2805857. PMID 19956261.
- ^ Donald, Voet (2011). Biochemistry. Voet, Judith G. (4th ed.). Hoboken, NJ: John Wiley & Sons. ISBN 9780470570951. OCLC 690489261.
- ^ Satir, Peter; Søren T. Christensen (26 March 2008). "Structure and function of mammalian cilia". Histochemistry and Cell Biology. 129 (6): 687–93. doi:10.1007/s00418-008-0416-9. PMC 2386530. PMID 18365235. 1432-119X.
- ^ a b c Varadi, Mihaly; Vranken, Wim; Guharoy, Mainak; Tompa, Peter (1 January 2015). "Computational approaches for inferring the functions of intrinsically disordered proteins". Frontiers in Molecular Biosciences. 2: 45. doi:10.3389/fmolb.2015.00045. PMC 4525029. PMID 26301226.
- ^ 단백질 앙상블 데이터베이스
- ^ Mittag, Tanja; Marsh, Joseph; Grishaev, Alexander; Orlicky, Stephen; Lin, Hong; Sicheri, Frank; Tyers, Mike; Forman-Kay, Julie D. (14 March 2010). "Structure/function implications in a dynamic complex of the intrinsically disordered Sic1 with the Cdc4 subunit of an SCF ubiquitin ligase". Structure. 18 (4): 494–506. doi:10.1016/j.str.2010.01.020. ISSN 1878-4186. PMC 2924144. PMID 20399186.
- ^ De Biasio, Alfredo; Ibáñez de Opakua, Alain; Cordeiro, Tiago N.; Villate, Maider; Merino, Nekane; Sibille, Nathalie; Lelli, Moreno; Diercks, Tammo; Bernadó, Pau (18 February 2014). "p15PAF is an intrinsically disordered protein with nonrandom structural preferences at sites of interaction with other proteins". Biophysical Journal. 106 (4): 865–874. Bibcode:2014BpJ...106..865D. doi:10.1016/j.bpj.2013.12.046. ISSN 1542-0086. PMC 3944474. PMID 24559989.
- ^ Kragelj, Jaka; Palencia, Andrés; Nanao, Max H.; Maurin, Damien; Bouvignies, Guillaume; Blackledge, Martin; Jensen, Malene Ringkjøbing (17 March 2015). "Structure and dynamics of the MKK7-JNK signaling complex". Proceedings of the National Academy of Sciences of the United States of America. 112 (11): 3409–3414. Bibcode:2015PNAS..112.3409K. doi:10.1073/pnas.1419528112. ISSN 1091-6490. PMC 4371970. PMID 25737554.
- ^ Allison, Jane R.; Rivers, Robert C.; Christodoulou, John C.; Vendruscolo, Michele; Dobson, Christopher M. (25 November 2014). "A relationship between the transient structure in the monomeric state and the aggregation propensities of α-synuclein and β-synuclein". Biochemistry. 53 (46): 7170–7183. doi:10.1021/bi5009326. ISSN 1520-4995. PMC 4245978. PMID 25389903.
- ^ Sivakolundu, Sivashankar G.; Bashford, Donald; Kriwacki, Richard W. (11 November 2005). "Disordered p27Kip1 exhibits intrinsic structure resembling the Cdk2/cyclin A-bound conformation". Journal of Molecular Biology. 353 (5): 1118–1128. doi:10.1016/j.jmb.2005.08.074. ISSN 0022-2836. PMID 16214166.
- ^ Zhang, Gong; Ignatova, Zoya (1 February 2011). "Folding at the birth of the nascent chain: coordinating translation with co-translational folding". Current Opinion in Structural Biology. 21 (1): 25–31. doi:10.1016/j.sbi.2010.10.008. ISSN 0959-440X. PMID 21111607.
- ^ Alberts, Bruce; Alexander Johnson; Julian Lewis; Martin Raff; Keith Roberts; Peter Walters (2002). "The Shape and Structure of Proteins". Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. ISBN 978-0-8153-3218-3.
- ^ Anfinsen, C. (1972). "The formation and stabilization of protein structure". Biochem. J. 128 (4): 737–49. doi:10.1042/bj1280737. PMC 1173893. PMID 4565129.
- ^ Jaenicke, R.; Heber, U.; Franks, F.; Chapman, D.; Griffin, Mary C. A.; Hvidt, A.; Cowan, D. A. (1990). "Protein Structure and Function at Low Temperatures [and Discussion]". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 326 (1237): 535–553. doi:10.1098/rstb.1990.0030. JSTOR 2398703. PMID 1969647.
- ^ Kendrew, J.C.; Bodo, G.; Dintzis, H. M.; Parrish, R. G.; Wyckoff, H.; Phillips, D.C. (1958). "A Three-Dimensional Model of the Myoglobin Molecule Obtained by X-Ray Analysis". Nature. 181 (4610): 662–666. Bibcode:1958Natur.181..662K. doi:10.1038/181662a0. PMID 13517261. S2CID 4162786.
- ^ Krimm S, Bandekar J (1986). "Vibrational spectroscopy and conformation of peptides, polypeptides, and proteins". Advances in Protein Chemistry Volume 38. Adv. Protein Chem. Advances in Protein Chemistry. Vol. 38. pp. 181–364. doi:10.1016/S0065-3233(08)60528-8. ISBN 9780120342389. PMID 3541539.
- ^ Lessing, J.; Roy, S.; Reppert, M.; Baer, M.; Marx, D.; Jansen, T.L.C.; Knoester, J.; Tokmakoff, A. (2012). "Identifying Residual Structure in Intrinsically Disordered Systems: A 2D IR Spectroscopic Study of the GVGXPGVG Peptide". J. Am. Chem. Soc. 134 (11): 5032–5035. doi:10.1021/ja2114135. hdl:11370/ff19c09b-088a-48f0-afee-2111a9b19252. PMID 22356513.
- ^ Jansen, T.L.C.; Knoester, J. (2008). "Two-dimensional infrared population transfer spectroscopy for enhancing structural markers of proteins". Biophys. J. 94 (5): 1818–1825. Bibcode:2008BpJ....94.1818J. doi:10.1529/biophysj.107.118851. PMC 2242754. PMID 17981904.
- ^ Minde DP, Maurice MM, Rüdiger SG (2012). "Determining biophysical protein stability in lysates by a fast proteolysis assay, FASTpp". PLOS ONE. 7 (10): e46147. Bibcode:2012PLoSO...746147M. doi:10.1371/journal.pone.0046147. PMC 3463568. PMID 23056252.
- ^ Kumari I, Sandhu P, Ahmed M, Akhter Y (August 2017). "Molecular Dynamics Simulations, Challenges and Opportunities: A Biologist's Prospective". Curr. Protein Pept. Sci. 18 (11): 1163–1179. doi:10.2174/1389203718666170622074741. PMID 28637405.
- ^ Laskowski, RA (2011). "Protein structure databases". Mol Biotechnol. 48 (2): 183–98. doi:10.1007/s12033-010-9372-4. PMID 21225378. S2CID 45184564.
- ^ Murzin, A. G.; Brenner, S.; Hubbard, T.; Chothia, C. (1995). "SCOP: A structural classification of proteins database for the investigation of sequences and structures" (PDF). Journal of Molecular Biology. 247 (4): 536–540. doi:10.1016/S0022-2836(05)80134-2. PMID 7723011. Archived from the original (PDF) on 26 April 2012.
- ^ Orengo, C. A.; Michie, A. D.; Jones, S.; Jones, D. T.; Swindells, M. B.; Thornton, J. M. (1997). "CATH--a hierarchic classification of protein domain structures". Structure. 5 (8): 1093–1108. doi:10.1016/S0969-2126(97)00260-8. PMID 9309224.
- ^ a b Pascual-García, A.; Abia, D.; Ortiz, A.R.; Bastolla, U. (2009). "Cross-over between discrete and continuous protein structure space: insights into automatic classification and networks of protein structures". PLOS Computational Biology. 5 (3): e1000331. Bibcode:2009PLSCB...5E0331P. doi:10.1371/journal.pcbi.1000331. PMC 2654728. PMID 19325884.
- ^ Holm, L; Rosenström, P (July 2010). "Dali server: conservation mapping in 3D". Nucleic Acids Research. 38 (Web Server issue): W545–9. doi:10.1093/nar/gkq366. PMC 2896194. PMID 20457744.
- ^ Zhang Y (2008). "Progress and challenges in protein structure prediction". Curr Opin Struct Biol. 18 (3): 342–348. doi:10.1016/j.sbi.2008.02.004. PMC 2680823. PMID 18436442.